【客户文献解读】IF=7.543,多组学联用结合互作技术确定RPS4Y1是VKH疾病中重要的CsA和CS抗性基因
信息来源:金开瑞 作者:genecreate 发布时间:2020-06-19 10:58:42
题目:Identification of Ribosomal Protein S4, Y-Linked 1 as a cyclosporin A plus corticosteroid resistance gene
期刊:Journal of Autoimmunity
影响因子(IF):7.543
研究背景
皮质类固醇(CS)联合环孢素A (CsA)被广泛用于治疗自身免疫性疾病、自身炎症性疾病和移植排斥反应。然而,一些患者对联合治疗方案没有反应或产生耐药性。在Vogt-Koyanagi-Harada (VKH)疾病模型中,通过转录组学(RNA-seq)、蛋白质组学(iTRAQ)体外检测等一系列实验,以筛选和验证潜在的耐药分子。研究发现在CsA、Cs耐药和-敏感的VKH患者来源的CD4+ T淋巴细胞中共有1697个差异表达基因(DEGs)和21个差异表达蛋白(DEPs)。核糖体蛋白S4和Y-Linked 1 (RPS4Y1)被证实调节男性VKH患者CD4+ T淋巴细胞对CsA和CS的抗性。作者发现CLB可以通过抑制RPS4Y1逆转抗性。综上所述,确定RPS4Y1是VKH疾病中重要的CsA和CS抗性基因。
研究内容及结果
1. CsA和CsA耐药及敏感VKH患者CD4+T细胞的转录和蛋白谱分析
作者应用RNA-seq技术检测来自12例耐药患者及19例敏感患者分离的CD4+T淋巴细胞mRNA表达水平的差异。与敏感患者相比,作者发现1697个差异表达耐药患者的基因(DEGs)(1.2倍变化,FDR<0.05)。其中822个基因上调表达,875个基因下调表达(图1A)。
作者还运用蛋白质组学(iTRAQ)开展了类似研究,发现与敏感患者相比,在耐药患者中发现21个差异表达蛋白(6个上调表达,15个下调表达(图1C))(DEPs)(1.2倍变化,p<0.05)。OmicsBean对DEGs和DEPs进行了功能分析(GO及KEGG) (http://www.omicsbean.cn)。DEGs 的GO分析结果显示:4604个生物过程(BP),499个细胞成分(CC),631个分子功能的功能通路被富集出来;此外26条KEGG通路被富集出来(图1B)。DEPs 的GO分析显示422个BPs,93个CCs,38个MFs及 13个KEGG功能通路被富集出来(图1D)。从GO和KEGG富集出来的通路可以看到,差异基因及蛋白主要是与炎症和免疫功能相关的,如NF-kappa B、抗原处理和递呈及MAPK等途径。

图1 耐药和敏感VKH患者CD4+T细胞的转录组和蛋白质组分析
(A) 火山图显示差异基因,与敏感患者相比,耐药患者的基因上调(红色)或下调(绿色)被认为是FDR<0.05,两者之间的倍数变化>1.2两组;
(B) GO和KEGG对DEGs的功能通路富集,p<0.05被认为是显著的富集;
(C) 火山图显示与敏感患者相比,耐药患者中的蛋白质显著上调(红色)或下调(绿色)的DEPs被认为是p<0.05,且两者之间的倍数变化>1.2两组;
(D) GO和KEGG对DEPs的富集,p<0.05为显著富集。DEPs:差异表达蛋白质。
2. 转录组与蛋白质组联合分析
通过对两个omics数据集(242个无注释)的综合分析作者发现:RPS4Y1和HLA-DQA1在mRNA和蛋白质水平上差异表达(图2A)。与敏感患者相比,基因及蛋白层面结果均显示RPS4Y1在耐药患者中上调,而HLA-DQA1下调(图2B)。此外,生物信息学分析显示两个候选基因参与了45个重要BPs,9个CCs,4个MFs和1重要的KEGG途径(图2C)。GO富集分析显示两个基因的详细功能分类并对其进行展示(图2D)。

图2 转录组与蛋白质组学联合分析
(A) vene图重叠部分显示候选分子RPS4Y1和HLA-DQA1在两个omics数据集。
(B) RPS4Y1和HLA-DQA1mRNAs与蛋白的相关性。
(C) 对RPS4Y1和HLA-DQA1进行富集分析,p<0.05为显著富集。
(D) GO富集的详细功能分类。
3. RPS4Y1是VKH病中一个潜在的CS a和CS抗性基因研究
针对多组学联合分析筛选到的两个基因,作者进一步通过RT-qPCR和PRM分别进行反向验证,检测结果显示:RPS4Y1和HLA-DQA1的蛋白表达趋势与转录组实验结果保持一致。RT-qPCR结果显示:RPS4Y1在男性耐药患者表达上调(图3A),PRM结果显示相同的表达模式(图3C和D)。另一方面,PRM结果显示耐药患者HLA-DQA1蛋白表达下降(图3C和D),与蛋白质组实验结果保持一致。RT-qPCR结果显示耐药与非耐药患者HLA-DQA1基因表达无显著性差异(P>0.05)。(图3B)。
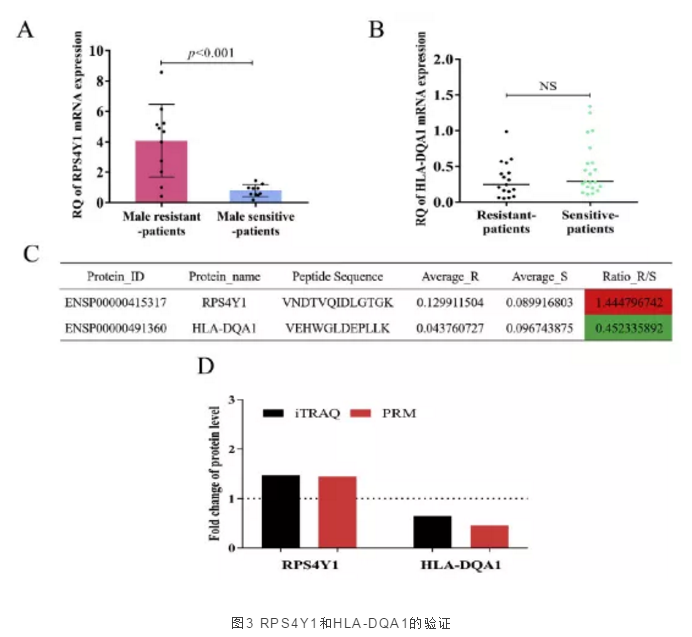
图3 RPS4Y1和HLA-DQA1的验证
(A和B)RT-qPCR法检测RPS4Y1(A)和HLA-DQA1
(B)的mRNA表达,GAPDH做内参对照,11例男性耐药,10例男性敏感患者检测RPS4Y1,17例耐药,21例敏感患者检测HLA-DQA1检测。(C) PRM用于验证蛋白质RPS4Y1(男性患者)和HLA-DQA1的水平,用R/S比值表示两组间蛋白水平的倍数变化,R表示耐药患者S代表敏感病人。
(B)的mRNA表达,GAPDH做内参对照,11例男性耐药,10例男性敏感患者检测RPS4Y1,17例耐药,21例敏感患者检测HLA-DQA1检测。(C) PRM用于验证蛋白质RPS4Y1(男性患者)和HLA-DQA1的水平,用R/S比值表示两组间蛋白水平的倍数变化,R表示耐药患者S代表敏感病人。
(D) iTRAQ与PRM结果的比较。
4. RPS4Y1对CD4+T细胞体外抗CsA和CS的调节作用
根据组学及验证结果,作者推测RPS4Y1可能对CsA及CS耐药性有一定的调节作用并进行功能检测实验进行验证。
通过转染过表达慢病毒感染CD4+T细胞(来源于5个男性敏感病人),同时构建干扰siRNA进行干扰实验转染CD4+T细胞(来源于8个男性耐药病人)。作者通过流式细胞术检测IL-17+和IFN-γ+CD4+T淋巴细胞,ELISA实验检测IL-17和IFN-γ细胞上清含量。结果显示CsA和Dex对IFN-γ+CD4+T细胞量无明显影响(图4A和B)。沉默RPS4Y1后CsA和Dex对细胞数量有下调影响。
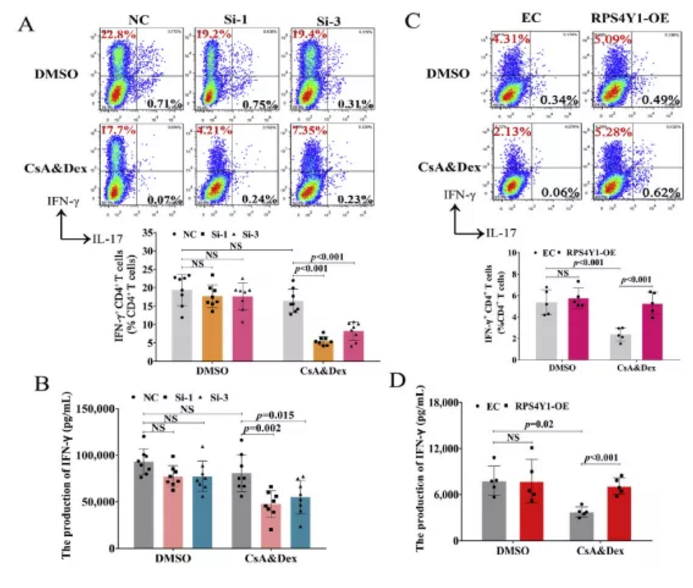
图4 RPS4Y1在体外调节CD4+T细胞对CsA和CS的抵抗
(A)流式细胞仪检测IL-17+和IFN-γ+CD4+T细胞占比;(B)ELISA法检测8例男性耐药患者CD4+T细胞培养上清液中IFN-γ的产生;
(C和D)(C)流式细胞检测IL-17+和IFN-γ+CD4+T细胞的细胞占比
(C)和(D)ELISA检测上清IFN-γ的含量。
5. CLB(苯丁酸氮芥)通过抑制RPS4Y1的表达进而抑制VKH患者对CsA和CS的耐药性
作者为探究CLB对耐药患者作用的机制探,对来自于9个男性抵抗者的CD4+T细胞进行不同方式的治疗分组:包括DMSO(对照)、CsA和Dex(处理组1)、CLB和CLB加CsA&Dex治疗(处理组2)。结果表明,与DMSO对照组相比,治疗组IFN-γ+CD4+T细胞占比明显升高;CLB加环孢素A和地塞米松治疗组降低,轻度CLB治疗组减少但无统计学意义(图5A)。同时,ELISA试验的结果也显示了同样的趋势(图5B)。与对照组相比,CsA和Dex药物处理组中IFN-γ+CD4+T细胞的数量占比及INF-γ的含量并没有受到影响(图5A和B)。IL-17+CD4+T细胞及IL-17的含量也没有显著性差异变化(图5A)。预示着CLB可能起着一个辅助的功能,并推测这种功能是由于抑制了RPS4Y1功能或者表达水平引起的。
作者进一步通过RT-qPCR检测证实与对照组相比,添加CLB药物处理后RPS4Y1的基因表达量下降,同时只添加CLB药物组的表达量最低(Fig.5C)。这种情况在蛋白层面同样得到了验证(Fig.5D)。作者同时观察到经CsA & Dex处理组样本中RPS4Y1基因及蛋白表达相对于DMSO对照组略微上调(Fig.5C 和D)。进一步通过挽救实验显示,CLB联合CsA、Dex治疗对IFN-γ+ CD4+ T占比率及IFN-γ的含量的抑制效果在加入RPS4Y1过表达后消失了(Fig5E-G)。综合以上结果显示:CLB通过抑制RPS4Y1的表达抵消VKH患者对CsA及CS的耐药性。
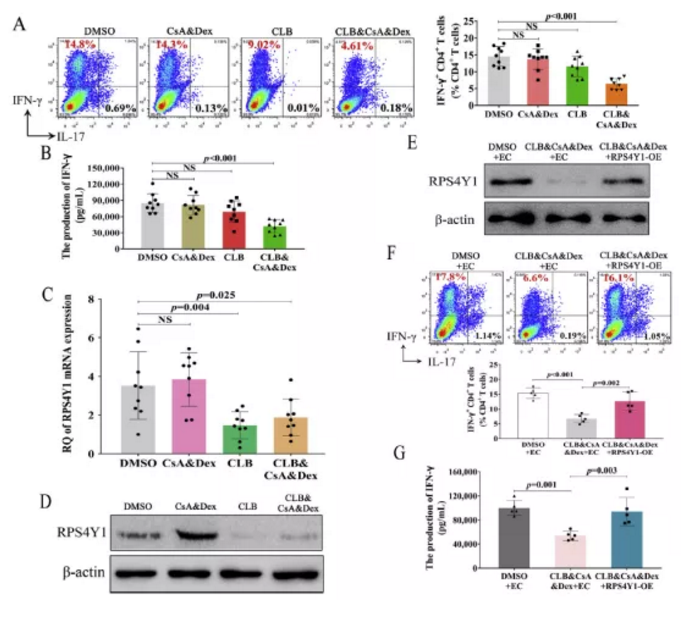
图5 CLB通过降低RPS4Y1的表达逆转VKH患者CD4+T细胞对CsA和CS的抗性。
(A)流式细胞检测IFN-γ+CD4+T细胞,(B)ELISA检测9例男性耐药患者经DMSO,CsA&Dex,CLB,CLB&CsA&Dex. 孵育后CD4+T细胞上清液中IFN-γ的产生
(C)RT-qPCR检测RPS4Y1的mRNA水平;(D)Western blot检测RPS4Y1的蛋白水平;
(E)Western blot检测RPS4Y1蛋白;
(F)流式细胞实验检测IFN-γ+CD4+T细胞的频率。
小结
作者确定RPS4Y1是CSa和CS抗性基因。并通过功能实验及挽救实验探索了关于RPS4Y1如何表达增加控制耐药。进一步的证据发现RPS4Y1在控制耐药性中的作用是通过对药物CLB辅助及RPS4Y1表达降低有关。作者的研究是专注于VKH疾病,但也可应用于其他环境CsA和CS抵抗,包括其他自身免疫性疾病或器官移植,为后续临床耐药基因的筛选及研究提供了鉴定的理论基础,具有极其重要的研究意义。
最新动态
-
04.22
一文读懂EMSA技术核心要点,让“emsa” 秒变“easy”
-
04.02
4·2世界孤独症日 | 聆听“星”声“泌”语,让爱来,让碍走
-
04.01
酵母杂交核心技术:深度Q&A帮你轻松突破实验瓶颈
-
03.19
【客户文献解读,IF>11】食管癌的"隐形推手":MALR-ILF3-HIF1a轴的强大作用
-
03.18
siRNA介绍及药物研发的现状前景
-
03.18
知无不“研”|5篇高分文献带你一览高通量酵母杂交的非凡魅力~
-
02.27
【客户文章分享】SHMT2 通过 5′UTR 依赖性 ADAM10 翻译启动介导小分子诱导的阿尔茨海默病病理学缓解过程
-
01.24
客户文献解读 | 中医为什么能治流感?是玄学还是运气?INT J NANOMED揭示鱼腥草抗病毒机制及范围!
-
01.24
客户文献分享,IF>11|Shank3:脑缺血再灌注损伤的守护者,揭示神经保护的新篇章
-
12.27
文献解读 | 高密度脂蛋白通过miR-181a-5p调控自噬影响血管新生










