那些发表CNS的大牛们,都在做什么?
信息来源:金开瑞 作者:genecreate 发布时间:2019-09-03 14:11:21
据统计,2014年开始,Hi-C相关的文章发表量呈指数级增长,其所发表的期刊,影响因子多集中于10-20分区域。历年影响因子大于10的文章可占据总文章数量的35%以上。2019年1-8月,Hi-C相关文章发表数量已达186篇。可见,开展Hi-C研究的工作力度在逐渐加强,Hi-C技术,也成为了发表10分以上文章的开挂神器。
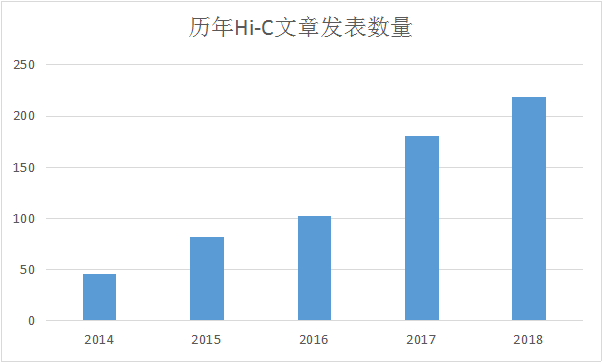
(图片自作,文章数量来源于Pubmed)
目前,Hi-C主要应用于揭示染色体区域(A/B compartments、TADs、Loops),协助理解基因的转录调控、疾病易感位点、基因组结构变异和表观遗传等方面。那么,在生物科研领域中到底如何运用?今天,小编特整理了几篇Hi-C的经典高分文献,带您详细了解一番。
LHX2- and LDB1-mediated trans interactions regulate olfactory receptor choice. Nature (IF=43.07). 2019 Jay.
研究人员借助Hi-C基因组测序技术,首次在哺乳动物的细胞核内发现一种巧妙的三维重排机制。
The Energetics and Physiological Impact of Cohesin Extrusion. Cell (IF=36.216). 2018 Sep.
作者围绕黏连蛋白挤压的能量和生理学影响进行了一系列研究,发现黏连蛋白挤压需要其内源性的ATP活性,大量的黏连蛋白结合在CTCF锚定位点附近产生了结构性的条纹;条纹结构可促进启动子-增强子的相互作用,而条纹锚定位点是肿瘤诱导TOP2β DNA断裂的主要位点。
Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosomeconformation capture. Nat Genet(IF= 25.455), 2018 May.
作者开发了一种Hi-C方法——DLO Hi-C,并利用这项技术成功的探索了基因组的三维结构和染色体易位。DLO Hi-C具有简单高效、高性价比、快速早期质控等优点。有助于探索全基因组的三维结构以及相关的研究。
The methyltransferase SETDB1 regulates a large neuron-specific topological chromatin domain. Nat Genet (IF=25.455). 2017 Aug.
作者通过细胞类型特异性3D基因组图谱,靶向表观基因组编辑等技术手段,识别出了一种能帮助基因组免受CTCF过度结合的SETDB1依赖性“盾牌”,揭示表观遗传对染色体关键结构域的影响。
3D Chromatin Structures of Mature Gametes and Structural Reprogramming during Mammalian Embryogenesis. Cell (IF=36.216). 2017 Jul.
该研究揭示了哺乳动物成熟精子和卵子的染色体3D结构及在早期胚胎发育过程中染色体结构的重编程变化,还首次发现染色体的高级结构与DNA甲基化的关联,并且发现早期发育的DNA去甲基化也与染色体的高级结构相关。
Allelic reprogramming of 3D chromatin architecture during early mammalian development. Nature (IF=43.07). 2017 Jul.
研究者对哺乳动物染色体三维结构在着床前胚胎发育过程中的动态重编程过程进行探讨,发现染色体的三维结构在受精后首先呈现出一种极其松散的状态,并在随后的胚胎早期发育过程中逐步地以亲本特异的方式建立和成熟。
3D structures of individual mammalian genomes studied by single-cell Hi-C. Nature (IF=43.07). 2017 Apr.
研究者运用单细胞Hi-C技术,研究了全基因组范围内整个染色质DNA在空间位置上的关系,首次在单细胞水平上复原哺乳动物细胞完整基因组3D结构图。
上述文章中,于2018年5国月发表于Nature Genetic上的DLO Hi-C技术,由金开瑞技术顾问、华中农业大学教授,曹罡课题组与华中农业大学李国亮教授课题组联合开发技术。
作为一种全新的染色体构象捕获技术,DLO Hi-C技术使全基因组染色体构象捕获实验的成本大大的降低,同时简化了实验步骤,使得实验成功率显著提高,对辅助基因组组装、解析基因组远程调控元件的功能、理解疾病易感位点以及检测染色体结构变异有着重要的意义。Nature Genetics同期发表专门评论性文章,对曹罡教授及其合作者的研究工作给予了较高评价。
目前,金开瑞联合技术顾问曹罡教授,特推出DLO Hi-C技术服务,为您解析基因组三维结构提供了一种新型、高效、经济的研究方案,让您的文章开启外挂。
DLO Hi-C服务流程:

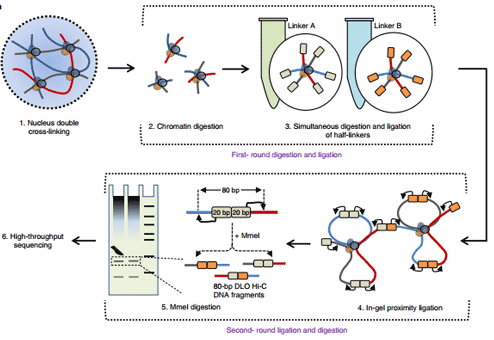
(图片来源:Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosomeconformation capture. Nat Genet, 2018 May.)
DLO Hi-C研究思路:
DLO Hi-C可以与RNA-Seq、ChIP-Seq、ATAC-Seq等数据进行联合分析,从基因调控网络和表观遗传网络来阐述生物体性状形成的相关机制。
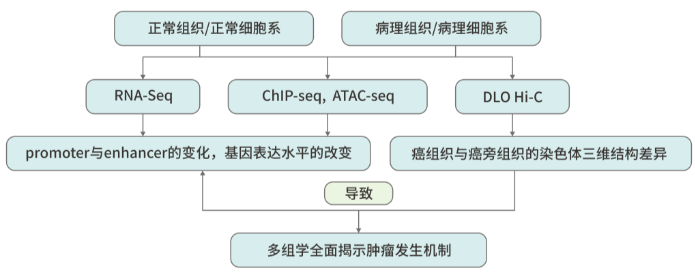
(图片自作)
DLO Hi-C技术优势:
- 微量细胞建库:正常建库与生信分析的样本量可低至10万个核
- 高成功率:细胞样本文库构建成功率几乎为100%
- 建库周期短:只需执行两轮简单的消化和连接步骤即可获得高质量的文库。
- 数据更准确:测序前质检,确保数据准确性
- 分辨率更高:在测序数据量更少的情况下,互作矩阵分辨率更高,染色质结构分析得到的数据也更多
- 较高的信噪比:使用多种措施来减少噪音,保证高质量的数据输出,分析更准确
- 量身定制个性化分析方案:提供DLO Hi-C的标准分析外,更注重与RNA-Seq、ChIP-Seq、ATAC-Seq和甲基化等多组学表观遗传分析,提供个性化的生信分析方案
最新动态
-
03.19
【客户文献解读,IF>11】食管癌的"隐形推手":MALR-ILF3-HIF1a轴的强大作用
-
03.18
siRNA介绍及药物研发的现状前景
-
03.18
知无不“研”|5篇高分文献带你一览高通量酵母杂交的非凡魅力~
-
02.27
【客户文章分享】SHMT2 通过 5′UTR 依赖性 ADAM10 翻译启动介导小分子诱导的阿尔茨海默病病理学缓解过程
-
01.24
客户文献解读 | 中医为什么能治流感?是玄学还是运气?INT J NANOMED揭示鱼腥草抗病毒机制及范围!
-
01.24
客户文献分享,IF>11|Shank3:脑缺血再灌注损伤的守护者,揭示神经保护的新篇章
-
12.27
文献解读 | 高密度脂蛋白通过miR-181a-5p调控自噬影响血管新生
-
12.27
16分+SCI【客户文章】Nature子刊:鞠怀强/徐瑞华团队揭示FGL1促进转移性结直肠癌免疫逃逸和进展的机制
-
12.13
Science子刊:肠道靶向纳米颗粒可针对产气荚膜梭菌感染提供特异性靶向抗菌肽
-
11.28
医生说:一天一个苹果 ,有没有理论依据?










