
三维基因组Hi-C
- 三维基因组
- Hi-C技术
- 染色体空间结构
- 相互作用网络
- 基因调控
服务特色
三维基因组Hi-C是一种高通量测序技术,用于研究染色体的空间结构和相互作用,深入了解基因组的三维折叠与调控机制。它在基因组学和疾病研究中扮演着重要角色,帮助揭示染色体间的相互作用网络及其与基因表达的关联。
服务介绍
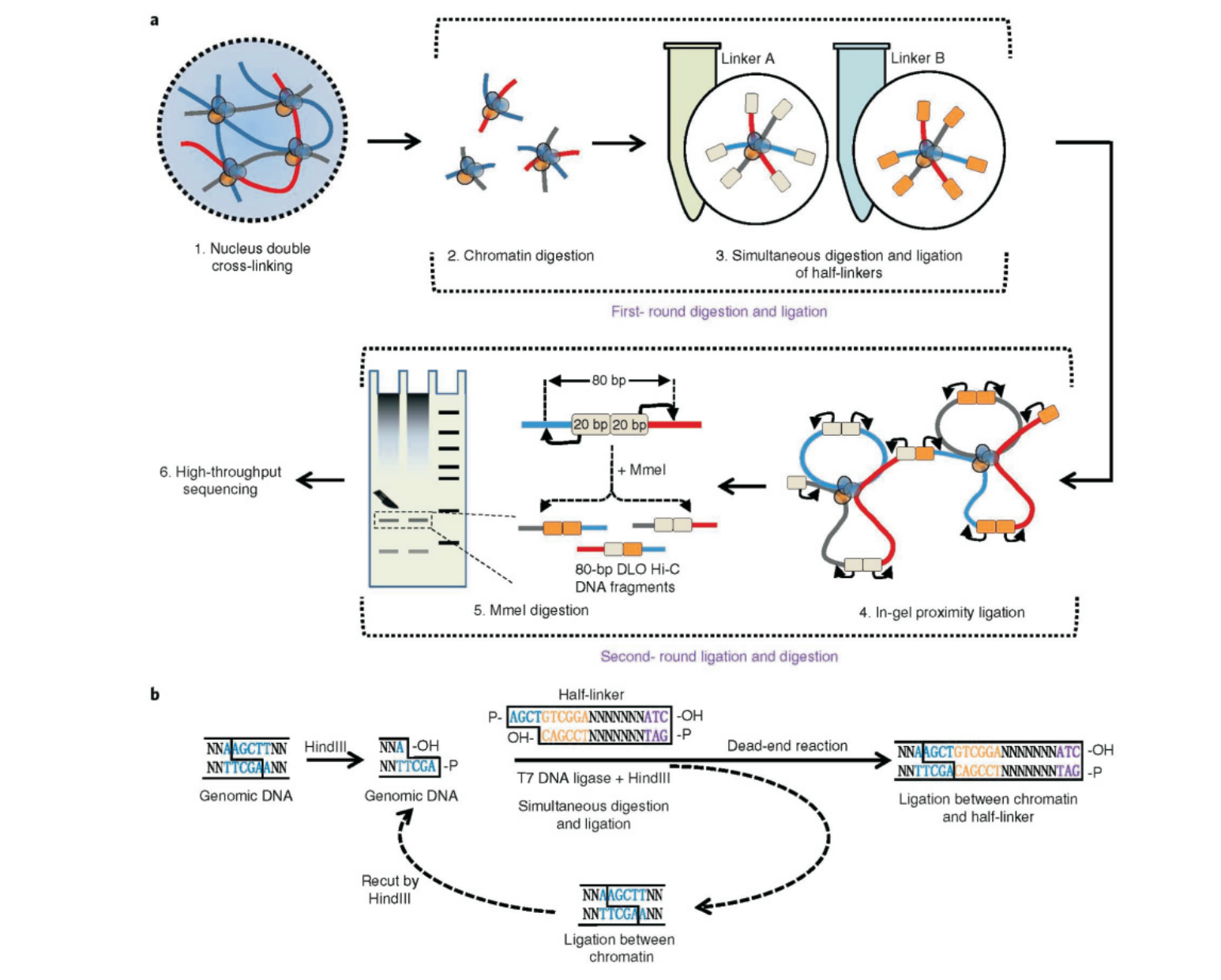
三维基因组学(Three-Dimensional Genomics, 3D Genomics),作为表观基因组学一部分,研究基因组的三维空间结构对基因组功能的影响。在细胞分化、组织发育、环境适应、疾病发生等生物学过程中都有染色质构象的调控。高通量染色质构象捕获技术Hi-C(High-throughput chromosome conformation capture),研究全基因组范围内染色质相互作用。Hi-C 通过染色质交联将空间构象固定,之后富集交联的 DNA 片段进行高通量测序,通过测序数据分析揭示染色质的远程相互作用以及空间结构,推导基因组的三维空间结构和可能的基因间的调控关系。
Hi-C实验步骤:交联——酶切——末端补平连接生物素——互作片段连接成环——通过生物素纯化DNA——上机测序。
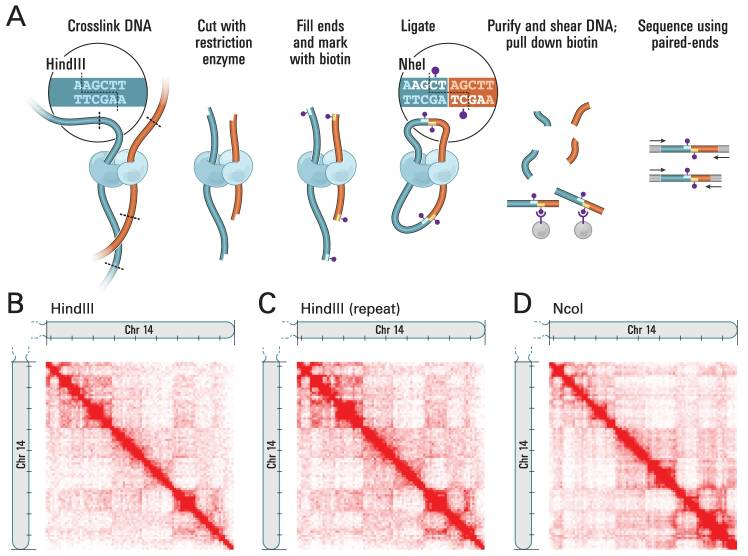
Figure 1 Hi-C实验示意(引用自Lieberman-Aiden et al., 2009)
经过测序后得到染色质互作信息文件,根据分析可以得到染色质高级结构,它可分为三种:AB Compartment、TAD和Loop。染色质根据转录活跃情况可以分为较活跃的A compartment和较不活跃的B compartment两种区室。在数十kb以及更高分辨率上,染色体沿对角线出现一个个三角形结构,这就是拓扑关联结构域(TAD),TAD内部之间相互作用频率显著高于与外部互作频率。大部分TAD是保守的,边界改变时可能改变基因互作情况。在10 kb以及更高分辨率上,会出现接触频率高于其邻域中的峰,峰反映了染色质环的存在,峰位点是染色质环的锚点。在转录因子或其他DNA结合蛋白介导下,增强子与启动子通过Loop在空间上相互接近,调控基因表达。
华中农业大学曹罡教授在Nature Genetic、Nature Communications等高水平期刊发表过多篇三维基因组学文章(Lin et al., 2018; Adeel et al., 2021; Lin et al., 2022),作为武汉金开瑞生物工程有限公司技术顾问为Hi-C实验与分析提供指导。
服务优势
- 全基因组覆盖:Hi-C技术能够对整个基因组进行调查,不受特定基因或区域的限制,为全面研究基因组的三维结构和相互作用提供了可能。
- 高分辨率:Hi-C技术具有较高的分辨率,能够检测到染色体区域间的相互作用频率,揭示基因组中不同区域的空间距离和相互关系。
- 无需先验信息:Hi-C技术不需要预先了解染色体相互作用的目标区域,因此可以发现新的基因组交互和调控元件,帮助发现潜在的功能区域。
- 高通量测序:Hi-C利用高通量测序技术,产生数百万个短的DNA序列读数,实现了大规模数据获取,提供了丰富的信息用于分析。
- 动态研究能力:Hi-C技术可以用于研究细胞分化、发育或响应刺激过程中染色体结构的动态变化,揭示基因组三维折叠的时空调控。
- 细胞异质性分析:Hi-C技术可以用于分析细胞群体中的异质性,发现细胞亚型和状态的差异,为理解细胞多样性提供信息。
服务流程
客户提供
与技术支持沟通除标准分析外分析内容,根据《武汉金开瑞表观项目送样指南》送样。目前只接受动物样本。
最终交付
- 标准分析内容:各个样本染色质互作数据文件、质控信息、AB compartment、TAD和Loop鉴定,以及各个样本之间Compartment、TAD和Loop差异。
- 多组学联合分析或其他个性化分析内容需事前讨论。
服务说明
| 项目 | 内容 |
| 建库、测序 | 每个样本生成2个文库,每个文库120GB测序量,每个样品产生不少于200GB raw data |
| 标准分析 | 质量控制、染色体顺式、反式互作、全基因组交互、A/B compartment分析、TAD分析 |
| 样本间差异A/B、TADs、Loops比对 | 商议 |
| Hi-C与RNA联合分析 | 商议 |
| 与其他组学联合分析 | 商议 |
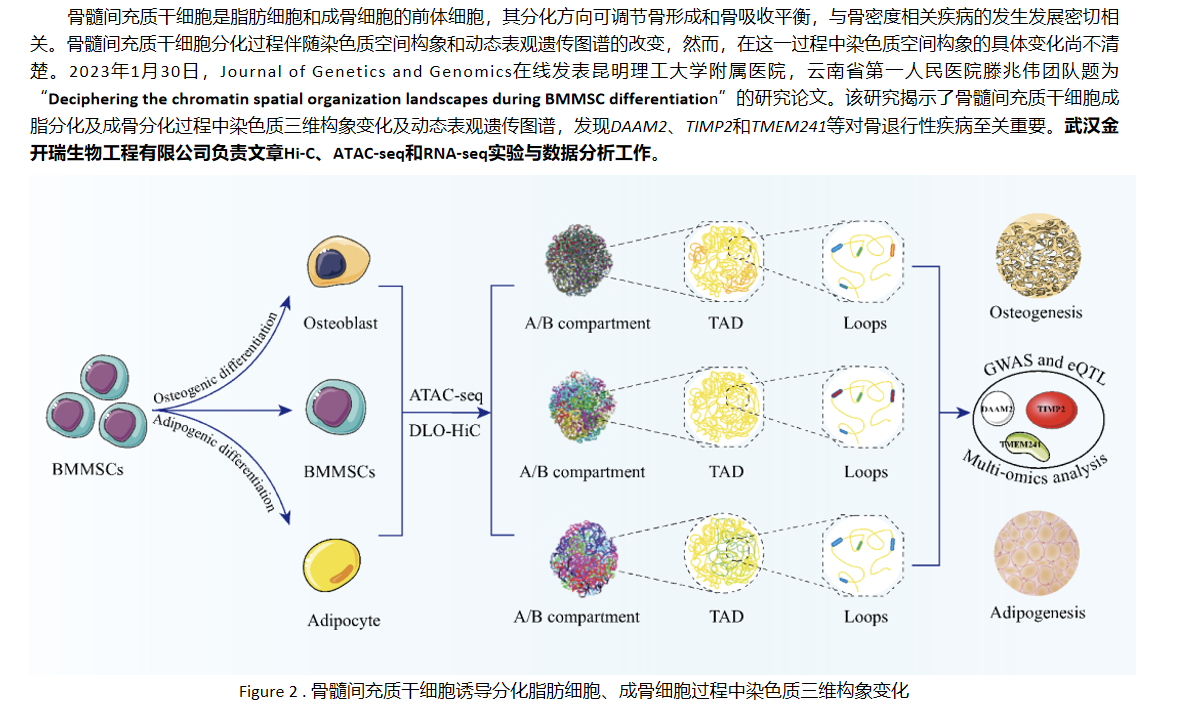
案例展示
相关资源
1、三维基因组Hi-C技术在基因组学研究中有着广泛的应用
● 染色体空间结构研究:Hi-C技术可以揭示染色体的三维结构和空间组织,如染色体环域(chromosome compartment)、TAD(染色质域)以及A/B亚染色质区域等,帮助我们了解染色体在细胞核内的折叠状态和整体结构。
● 染色体相互作用网络构建:Hi-C可以构建染色体之间的相互作用网络,研究不同染色体区域之间的相互关系和距离,探索基因组中的空间相互作用网络。
● 基因调控研究:Hi-C可以揭示染色体区域之间的相互作用,帮助鉴定可能的增强子与靶基因之间的相互作用,深入了解基因调控的机制。
● 细胞分化与发育:Hi-C可以用于跟踪细胞分化和发育过程中的染色体结构和相互作用的动态变化,帮助理解细胞命运决定和特化的分子机制。
● 疾病研究:Hi-C可以用于研究染色体异常、重排、基因缺失等与疾病相关的基因组变化,发现与疾病发病机制相关的信息。
● 药物靶点发现:Hi-C可以用于评估药物对染色体结构和相互作用的影响,帮助筛选潜在的药物靶点或开发个性化治疗策略。
● 细胞异质性分析:Hi-C可以用于分析细胞群体中的异质性,鉴别不同细胞亚型或状态,帮助理解细胞多样性。
2、Hi-C数据库资源和网站:
● 3DGenomes Database:
3DGenomes数据库提供了大量来自不同生物种类和细胞类型的Hi-C数据。该数据库支持Hi-C数据的浏览、查询和交互式可视化,方便研究者进行数据挖掘和分析。(www.3dgenomesdb.org)
● ENCODE Hi-C Data Portal:
ENCODE是一个重要的国际合作项目,其Hi-C Data Portal提供了大量的Hi-C数据和相关的实验信息。用户可以通过该网站获取不同细胞类型和条件下的Hi-C数据。(www.encodeproject.org/data-standards/hic/)
● GEO - Gene Expression Omnibus:
GEO是一个公共数据库,提供了许多Hi-C数据集,包括原始的测序数据和已经分析过的结果。可以通过关键词搜索或浏览数据集来获取感兴趣的Hi-C数据。(www.ncbi.nlm.nih.gov/geo/)
● HiGlass:
HiGlass是一个交互式的Hi-C数据可视化工具,可以用于浏览和比较多个Hi-C数据集,并与其他组学数据进行整合展示。(higlass.io/)
● Juicebox:
Juicebox是一个用于Hi-C数据交互式分析和可视化的工具。它支持数据的加载、浏览、交互、比较和生成Hi-C相互作用图。(www.aidenlab.org/juicebox/)
● HiCExplorer:
HiCExplorer是一个开源的Hi-C数据分析工具,提供了多种功能,包括数据预处理、相互作用频率计算、TAD检测等。(hicexplorer.readthedocs.io/)
● 4DGenome:
4DGenome是一个整合了多种基因组三维结构和表观遗传数据的平台。它提供了多种交互式工具和可视化功能,用于探索基因组空间组织与基因调控的关系。(4dgenome.research.chop.edu/)
3、Hi-C技术可以根据实验样本和具体应用的不同,分为多种变种和改进版本,以下是一些常见的Hi-C技术的分类:
● 常规Hi-C: 常规Hi-C是最早的Hi-C技术,适用于大量细胞的测序,通过对整个细胞群体的染色体交联产物进行测序,揭示染色体的三维结构和相互作用网络。
● 单细胞Hi-C: 单细胞Hi-C是对单个细胞进行Hi-C测序的技术,它允许我们探索细胞间的异质性和个体间的差异,了解细胞类型和发育过程中的稳定或动态的三维结构。
● HiChIP: HiChIP是一种将染色体免疫沉淀和Hi-C技术相结合的方法,用于研究蛋白质与染色体的相互作用和调控网络。
● 4C-seq: 4C-seq是一种用于研究特定基因座相互作用的Hi-C变体,通过选择性扩增特定靶点附近的染色体相互作用产物,揭示特定基因与增强子之间的相互作用。
● 5C: 5C是一种用于定量检测特定基因座与其他基因座之间的相互作用频率的Hi-C技术,广泛应用于基因调控研究。
● In situ Hi-C: In situ Hi-C是一种在原位进行交联和测序的Hi-C技术,它可以更准确地反映染色体的真实三维结构。
● Capture Hi-C: Capture Hi-C是一种通过卡片化(capture)特定基因组区域的Hi-C技术,用于研究特定基因座与整个基因组的相互作用。
除了以上列举的常见Hi-C技术,还有许多其他改进版本和变体,这些技术不断发展和改进,为我们深入研究基因组的三维结构和调控机制提供了更多的可能性和选择。










