ChIP-seq
- 三次质控
- 自用抗体平台
- 项目经验丰富
服务特色
ChIP-seq是目前最成熟的蛋白质与DNA互作研究方法,金开瑞自有抗体平台、检测分析平台,前后三次质控,可最大限度的保障实验成果率。
服务介绍
ChIP-seq(Chromatin ImmunoPrecipitation Sequencing)是染色质免疫共沉淀和第二代高通量测序技术的结合,通过组蛋白/转录因子的特异性抗体,将特定的蛋白与DNA复合物沉淀下来。从而获取结合的DNA片段,再通过高通量测序技术,即可获得相应的片段信息。
服务优势
- 项目前后三次质控,保障实验成功率;
- 金开瑞自有抗体生产团队,选择抗体经验丰富;
- 细胞、植物、动物都有大量经验。
服务流程
客户提供
根据《武汉金开瑞表观项目送样指南》提供实验的样品,并提供ChIP级别抗体(公司可以提供选购意见)。
样品信息单:需详细填写样品来源、含量、状态及其他基本信息。
最终交付
- 项目报告与数据:
- 测序数据质量评估:过滤掉低质量数据,保证数据质量
- 与参考基因组比对:reads分布及比对结果可视化
- peak峰calling:分析蛋白结合位点
- motif分析:蛋白结合序列的偏好性
- peak峰相关基因注释:寻找蛋白潜在调控基因
- 差异peak分析:分析不同样本间差异peak峰
- 相关基因功能分析:相关基因GO, KEGG富集分析
服务说明
|
产品 |
测序数据量 |
平台 |
周期(自然日,提取+测序分析) |
备注 |
|
组蛋白ChIP-seq |
8G |
illumina |
45 |
需要组间比较时,每组至少两个生物学重复,每个样本会进行ChIP和Input测序 |
|
转录因子ChIP-seq |
6G |
illumina |
45 |
需要组间比较时,每组至少两个生物学重复,每个样本会进行ChIP和Input测序 |
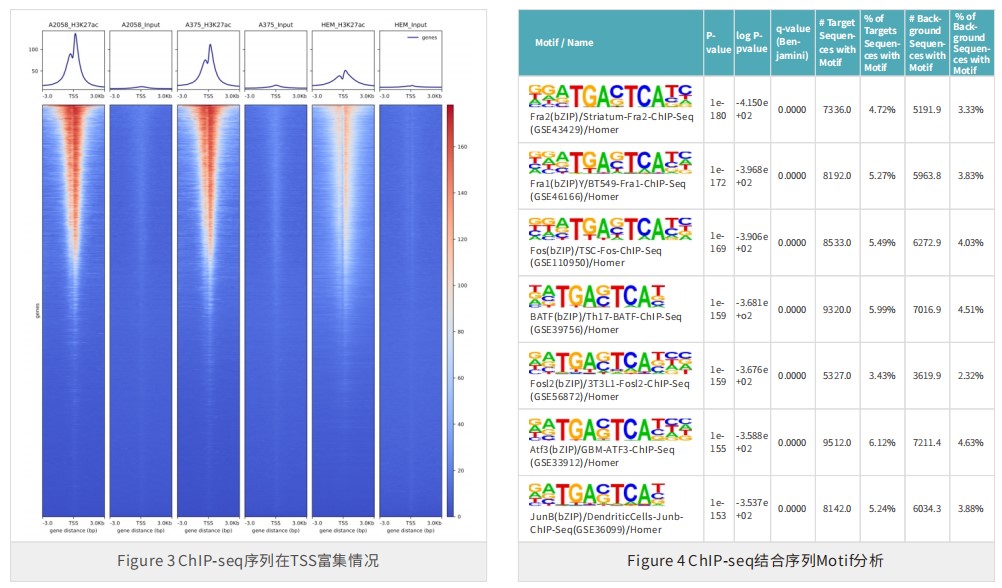
案例展示
常见问题与解析 (Q&A)
通常是≥10ng。随着目前建库技术的不断完善,低至5ng的DNA量也能被建库成功;少数测序公司为了降低自身风险提出20ng的要求,通过可以协商降低该送样要求。尤其是一些转录因子,最终能够捕获的DNA量会很低。
通常是≥10ng。随着目前建库技术的不断完善,低至5ng的DNA量也能被建库成功;少数测序公司为了降低自身风险提出20ng的要求,通过可以协商降低该送样要求。尤其是一些转录因子,最终能够捕获的DNA量会很低。
如果ChIP下来的DNA样品量非常少,在样品制备过程中通常需要经过一步PCR扩增的过程,PCR扩增主要是为了获得足够上机反应的DNA量。如果客户能够提供足够量的DNA样品,那就不需要再进行PCR扩增。由于是线性扩增,扩增前后的结果很相似,基本上不会影响测序结果。抗体的质量,特异性,ChIP的实验操作,实验设计方法,DNA片段长度范围,测序通量和质量等都会影响ChIP-Seq的结果。
(1)染色质开放程度的不均一性、DNA打碎方法、PCR扩增偏向性、基因组的重复程度以及测序和序列比对过程中的错误都会引入系统误差造成假阳性,测序后首先将序列比对到已知基因组上并确立真正的结合位点。 (2)对于转录因子,要寻找“峰”对应的下游调控基因(靶基因),或者构建转录因子结合位点的保守结合序列,如果转录因子的motif是已知的,则可以计算“峰”序列中包含motif序列的百分比,间接估计实验结果的可靠性。
相关资源
1、ChIP-seq技术的实验步骤:
● 交联与裂解:交联细胞或组织中蛋白质与DNA的相互作用,然后裂解细胞核,释放染色质。
● 免疫沉淀:使用特定抗体对目标蛋白质进行免疫沉淀,富集与该蛋白质结合的染色质片段。
● 反交联与DNA纯化:解除交联,将富集的染色质片段纯化出来。
● 文库构建:对富集的DNA片段进行文库构建,将其转化为可测序的DNA文库。
● 高通量测序:对构建好的DNA文库进行高通量测序,产生数百万个短的DNA序列读数。
● 数据分析:将测序数据进行比对,将序列读数映射到参考基因组上,并根据比对结果寻找与特定蛋白质结合的染色质区域。
2、ChIP-seq技术在基因组学研究中有着广泛的应用,包括但不限于:
● 转录因子结合位点鉴定:ChIP-seq可以帮助鉴定转录因子与基因组上的结合位点,揭示转录因子对基因调控的作用。
● 组蛋白修饰分析:ChIP-seq可以用于检测不同组蛋白修饰在基因组上的分布,如甲基化、乙酰化等,揭示染色质状态和基因表达的调控。
● 基因调控网络研究:ChIP-seq可以帮助构建基因调控网络,找出转录因子和其他调控因子与靶基因的相互作用关系。
● 疾病研究:ChIP-seq可以用于研究特定蛋白质在疾病发生和发展中的作用,如癌症等。
● 药物靶点发现:ChIP-seq可以帮助鉴定新的药物靶点,为药物研发提供新的目标。
3、ChIP-seq数据库资源和网站:
● ENCODE ChIP-seq Data Portal:
ENCODE是一个重要的国际合作项目,其ChIP-seq Data Portal提供了大量的ChIP-seq数据和相关的实验信息。用户可以通过该网站获取不同细胞类型和条件下的ChIP-seq数据。(encodeproject.org/data-standards/chip-seq/)
● GEO - Gene Expression Omnibus:
GEO是一个公共数据库,提供了许多ChIP-seq数据集,包括原始的测序数据和已经分析过的结果。可以通过关键词搜索或浏览数据集来获取感兴趣的ChIP-seq数据。(ncbi.nlm.nih.gov/geo/)
● ChIP-Atlas:
ChIP-Atlas是一个综合性的ChIP-seq数据库,提供了广泛的ChIP-seq数据集和相关的元数据信息。它支持ChIP-seq数据的交互式可视化和数据下载。(chip-atlas.org)
● Cistrome Data Browser:
Cistrome Data Browser是一个基因组调控数据的综合性数据库,包括ChIP-seq数据和相关的基因表达数据,可以用于基因调控网络的研究。(cistrome.org/db/#/)
● UCSC Genome Browser:
UCSC Genome Browser是一个广泛使用的基因组浏览器,它提供了许多公开共享的ChIP-seq数据集,并且可以与其他组学数据进行整合展示。(genome.ucsc.edu/)
● ChIPseeker:
ChIPseeker是一个R包,用于ChIP-seq数据的注释和可视化,可以帮助研究者解释和分析ChIP-seq结果。(bioconductor.org/packages/release/bioc/html/ChIPseeker.html)











