miRNA可以与mRNA结合调节体内基因的表达,通过影响蛋⽩的表达、修饰、酶活性等来调节⽣命活动的进⾏。lncRNA作为体内的ceRNA,可以与miRNA结合起到调节基因表达的作⽤。研究lncRNA与miRNA的相互作⽤对于研究⽣命具有重要的意义。
双荧光素酶检测是转录调控研究中⼗分重要的实验⼿段,主要应⽤于启动⼦和转录因⼦,以及miRNA和其靶基因互作的验证。
- 在双荧光素酶检测中,将萤⽕⾍荧光素酶作为实验报告基因,海肾荧光素酶作为对照报告基因。实验报告基因⽤于测试实验条件下基因的表达,⽽对照报告基因作为内对照。
- 以荧光素为底物来检测萤⽕⾍荧光素酶活性,荧光素酶可以催化荧光素,在荧光素氧化的过程中会发出⽣物荧光,然后通过化学发光仪测定。
● 转录因⼦与启动⼦
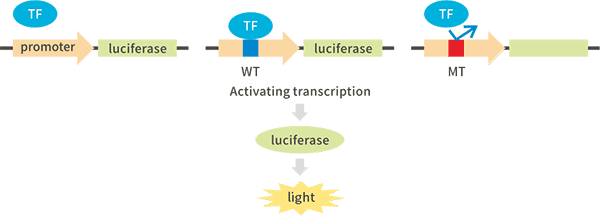
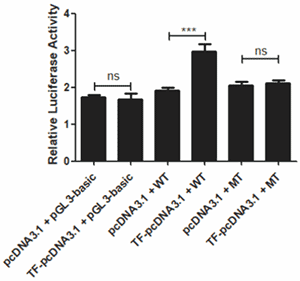
将启动子序列构建到萤火虫荧光素酶基因前,同时过表达转录因子。当转录因子与启动子上特异结合位点结合后激活荧光素酶基因转录,使萤火虫荧光素酶得以表达,最终荧光强度上升;当结合位点被突变后,转录因子无法与启动子结合,因此荧光值无明显变化。
● miRNA与靶基因
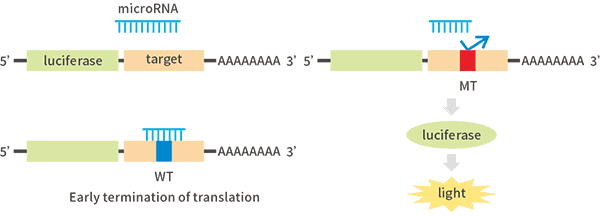
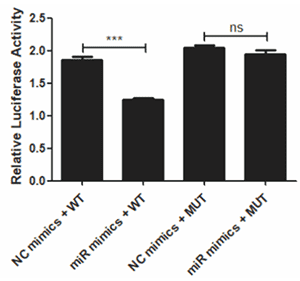
将靶基因序列构建到萤火虫荧光素酶基因3'区域,同时过表达microRNA。当microRNA与靶基 因上特异结合位点结合后,将干扰荧光素酶mRNA的翻译或导致其迅速降解,使荧光强度降低;当结合位点被突变后,microRNA无法与靶基因结合,因此荧光值无明显变化。
YY1-mediated reticulocalbin-2 upregulation promotes the hepatocellular carcinoma progression via activating MYC signaling.
期刊:Am J Cancer Res IF:5.177
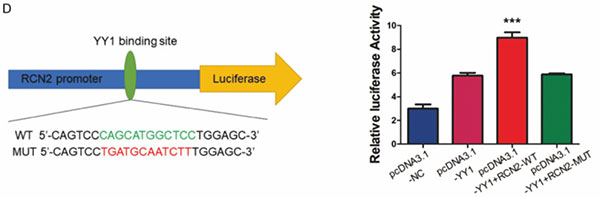
Schematic illustration of YY1 binding site on the wild type and mutant RCN2 promotor. Fluorescence activity in Huh7 cells with RCN2 wildtype and mutant promotor with YY1 pcDNA3.1.
蛋白与核酸的相互作用广泛存在于生命活动的调节中。基因的复制、转录、翻译、修饰等过程都离不开核酸和蛋白的互作。金开瑞提供RNA pull down+质谱、双荧光素酶、染色质免疫共沉淀、凝胶迁移或电泳迁移率检测(EMSA)、酵母单杂交、DNA pull down等技术来检测DNA/RNA与蛋白的相互作用。
凝胶迁移或电泳迁移率检测(Electrophoretic Mobility Shift Assay,EMSA)是一种检测蛋白质和DNA 序列相互结合的技术,可用于定性和定量分析。目前已用于研究DNA 结合蛋白和特定的DNA 序列的相互作用,是转录因子研究的经典方法。
金开瑞提供EMSA 检测技术服务,帮助您检测DNA 结合蛋白、RNA 结合蛋白、特定的蛋白质。
目的蛋白与生物素标记的DNA探针结合,电泳时蛋白-DNA复合物比无蛋白结合的DNA探针在凝胶中泳动的速度慢,显影出现相对滞后条带。
Functional Analysis of IRF1 Reveals its Role in the Activation of the Type I IFN Pathway in Golden Pompano, Trachinotus ovatus (Linnaeus 1758).
期刊:Int J Mol Sci. IF:4.183
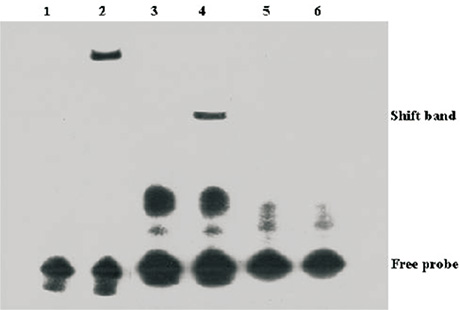
Binding reactions of IRF1 and ToIFNa3 prompter. Biotin-labeled EMSA probes were incubated with lysates of HEK293T cells containing ToIRF1 protein.WT,wild-type probe;MT:mutated probe.1,negative control;2,positive control;3,plus ToIFNa3-P2-WT5;4,ToIFNa3-P2-WT5 plus ToIRF1-Flag;5,plus ToIFNa3-P2-MT5;6,ToIFNa3-P2-MT5 plusToIRF1-Flag.
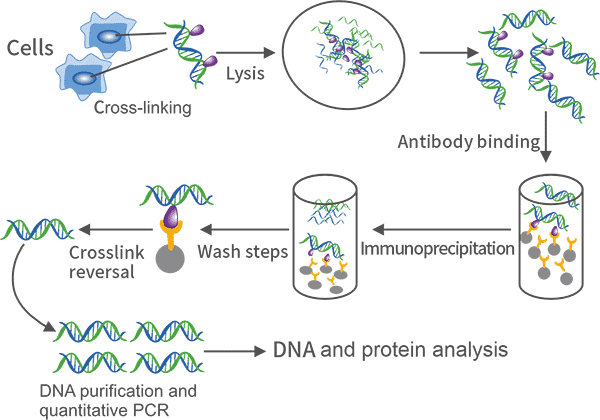
ChIP染色体免疫共沉淀(Chromatin Immunoprecipitation,ChIP)是用来研究蛋白质与DNA是否在体内存在相互作用,这项技术帮助研究者判断在细胞核中基因组的某一特定位置会出现何种组蛋白修饰。利用抗体抗原特异性结合,将与目的蛋白相结合的DNA片段沉淀下来,能够真实地反映结合在DNA序列上的调控蛋白。
YAP inhibits autophagy and promotes progression of colorectal cancer via upregulating Bcl-2 expression
期刊:Cell Death Dis IF:6.304
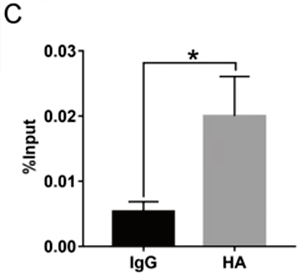
C ChIP-qPCR was performed in SW620 cells transfected with pcDNA3.1-HA-YAP to identify the enrichment of HA-YAP onto Bcl-2 promoter region. IgG served as an antibody control.
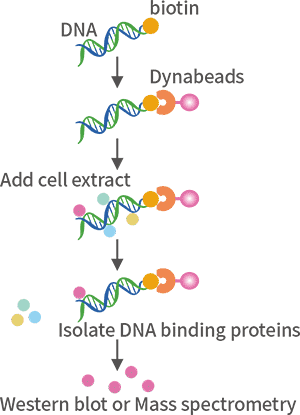
DNA pull down是用于分析蛋白质与DNA互作的一种分析技术。一般来说,该实验首先需要针对待研究目的基因的调控区域设计并制备特异性探针(以脱硫生物素标记为主流);同时,制备细胞核提取物;将探针和核提取物共同孵育,DNA结合蛋白就会和靶向序列特异性结合;然后,通过亲和素磁珠纯化蛋白质DNA复合物;最后针对获得的蛋白,使用WB验证或者质谱方式鉴定蛋白质类型。
应用:寻找已知的启动子序列所结合的未知蛋白(转录因子)
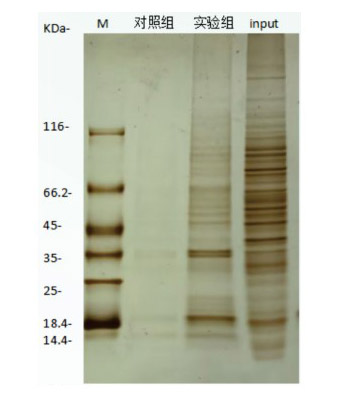
结果:实验组和对照组之间存在差异条带
酵母单杂交技术是在酵母双杂基础上发展而来的一种研究核酸-蛋白相互作用的工具,被广泛用于研究真核细胞内基因的表达调控,如鉴别DNA结合位点发现潜在的结合蛋白基因、分析DNA结合结构域信息等。
| 项目名称 | 应用 | 互补实验 |
|---|---|---|
| 酵母杂交文库构建 | 酵母杂交筛选 | / |
| 酵母单/双杂交验证 | 已知蛋白与蛋白/启动子之间的互作 | EMSA,双荧光素酶,Co-IP,GST pulldown |
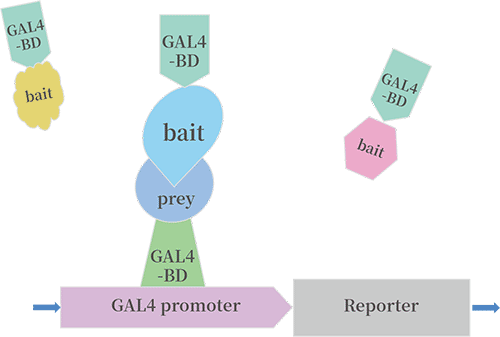
核酵母单/双杂交系统原理:酵母转录因子Gal4包含DNA结合结构域(Gal4-BD)和转录激活域(Gal4-AD),诱饵(prey)与Gal4-BD融合,猎物(bait)蛋白与Gal4-AD融合,当诱饵和猎物发生相互作用时,Gal4-BD和Gal4-AD在空间上充分接近,可呈现完整的GAL4转录因子活性,启动激活报告基因AUR1-C、ADE2、HIS3和MEL1的转录。

1: Pabai-Bait+PGADT7(自激活验证)
2: Pabai-Bait+PGADT7-Prey(实验组)
+: Pabai-P53+PGADT7-53(阳性对照)
- : Pabai-P53+PGADT7(阴性对照)
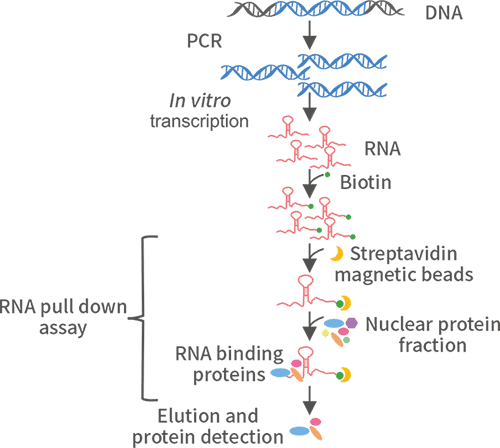
蛋白质与RNA 的相互作用是许多细胞功能的核心,如蛋白质合成、mRNA 组装、病毒复制、细胞发育调控等。使用体外转录法标记生物素RNA 探针,然后与胞浆蛋白提取液孵育,形成RNA-蛋白质复合物。该复合物可与链霉亲和素标记的磁珠结合,从而与孵育液中的其他成分分离。复合物洗脱后,通过WB 实验检测特定的RNA 结合蛋白是否与RNA 相互作用。若待检测目的蛋白明确,选择WB鉴定;若不明确,则可选择质谱鉴定。
金开瑞现提供RNA pull down 检测技术服务, 使用特异性二抗进行检测,能有效避免抗体重链对结果的信号干扰。并且金开瑞配套自己的质谱平台,能显著提升检测效果。
Circular RNA circEsyt2 regulates vascular smooth muscle cell remodeling via splicing regulation
期刊:J Clin Invest IF:11.864
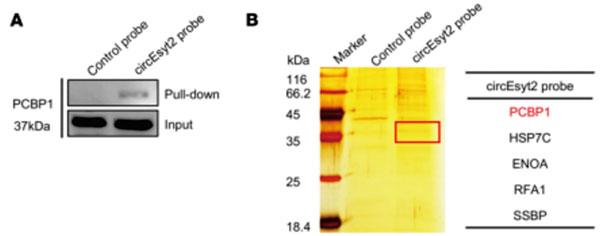
CircEsyt2 inhibits the nuclear trafficking of PCBP1 by binding directly to PCBP1. (A) Western blotting of proteins pulled down by control and circEsyt2 probes in circEsyt2-OE HEK293T cells using the PCBP1 antibody. (B) Identification of circEsyt2-binding proteins. Left: silver staining of pulled-down proteins in circEsyt2-OE HEK293T cells. Right: mass spectrometry showing the main proteins pulled down by the circEsyt2 probe.
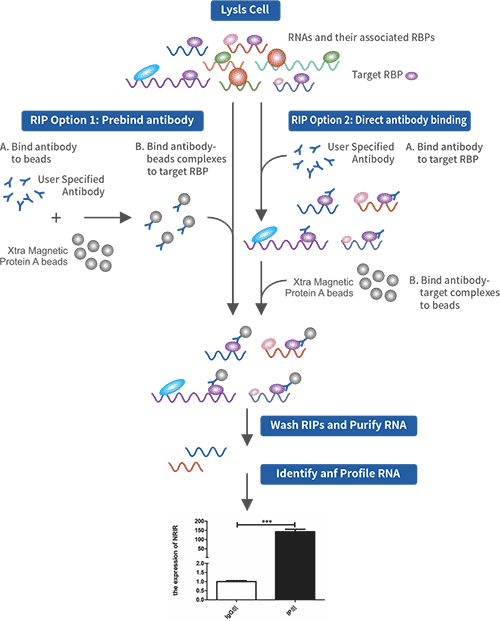
RIP 技术(RNA Binding Protein Immunoprecipitation Assay,RNA 结合蛋白免疫沉淀)主要是运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA 进行q-PCR验证或者测序分析。RIP 是研究细胞内RNA 与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工具,可以帮助我们发现miRNA 的调节靶点。
根据客户需求,金开瑞可以提供RNA/蛋白、RNA/RNA互作验证RIP技术服务。
CircPVT1 promotes proliferation of lung squamous cell carcinoma by binding to miR-30d/e
期刊:J Exp Clin Cancer Res IF:11.161
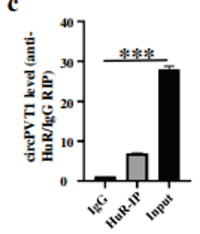
c RIP confirmed the relationship between circPVT1 and HuR in H520 cells. GAPDH mRNA was used as a non-HuR target control.
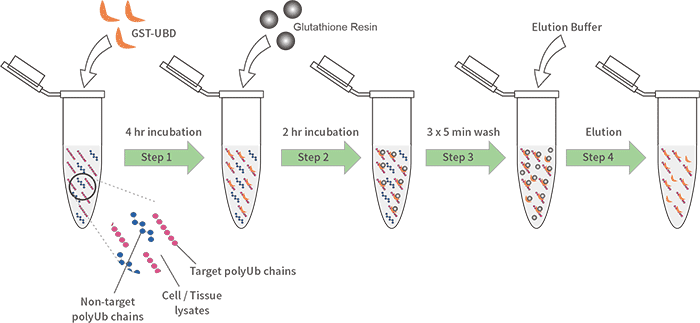
利用带有标签的已知蛋白作为诱饵蛋白(最常用的是GST标签,即谷胱甘肽巯基转移酶,glutathione S-transferase),诱饵蛋白特异性结合谷胱甘肽亲和树脂,当目的蛋白溶液过柱时,将诱饵蛋白及其相互作用的蛋白复合物捕获。复合物洗脱后,通过蛋白凝胶电泳分析两种蛋白间的相互作用,或者筛选相应的目的蛋白。
Aberrant oligodendroglial LDL receptor orchestrates demyelination in chronic cerebral ischemia
期刊:J Clin Invest IF:11.864
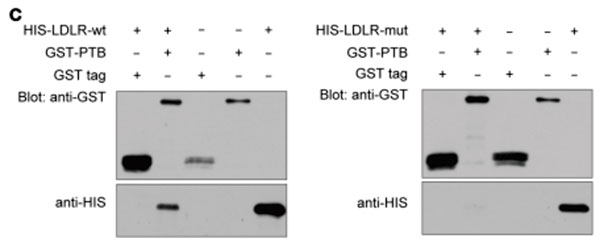
(C) GST pull-down assay revealing the interaction between LDLR and Shc (n = 3 in each group).
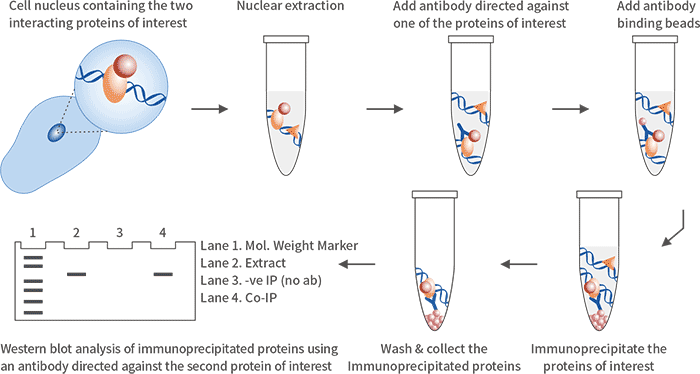
免疫共沉淀技术(co-Immunoprecipitation, co-IP)是一种研究两种蛋白在体内是否存在相互作用的有效方法,通过抗体和已知蛋白结合,从而捕获整个已知蛋白复合物,进而研究复合物中与已知蛋白存在相互作用的蛋白。
Aberrant oligodendroglial LDL receptor orchestrates demyelination in chronic cerebral ischemia
期刊:J Clin Invest IF:11.864
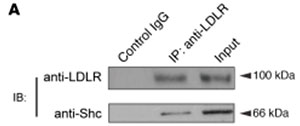
LDLR protects against demyelination by binding Shc and activating downstream MEK/ERK signaling. (A) Immunoassay of lysates of cerebral CC tissue after immunoprecipitation with LDLR, analyzed by immunoblotting (IB) with anti-LDLR and anti-Shc (n = 3 in each group).
酵母双杂交及系统是一种鉴定和检测蛋白质相互作用的研究方法,因其具有灵敏性高、功能强大、适用范围广等特点,现已被应用于多个研究领域。
金开瑞可提供酵母杂交文库构建、核酵母双杂交筛选、膜酵母双杂交筛选、酵母单/双杂交验证服务。
| 项目名称 | 应用 | 互补实验 |
|---|---|---|
| 酵母杂交文库构建 | 酵母杂交筛选 | / |
| 核酵母双杂交筛选 | 获得与已知蛋白互作的未知蛋白 | COIP,GST pulldown |
| 膜酵母双杂交筛选 | 研究膜蛋白之间的互作 | COIP,GST pulldown |
| 酵母单/双杂交验证 | 已知蛋白与蛋白/启动子之间的互作 | EMSA,双荧光素酶,COIP,GST pulldown |
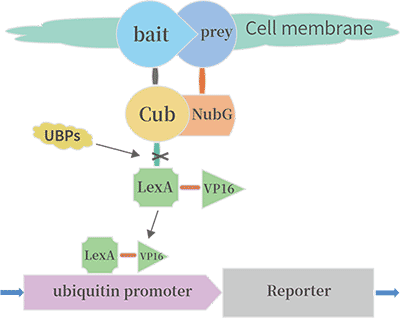
膜酵母双杂交系统原理:泛素ubiquitin包含N 端(Nub),C 端(Cub)结构域。首先,人为的将泛素 Nub的第3位异亮氨酸突变为甘氨酸(NubI 突变为 NubG)。这样与 Cub 的亲合力大大降低,避免了 Cub 和 Nub 自我结合或接近的可能性。在Cub结构域融合LexA-VP16 转录激活因子形成Cub-LexA-VP16。诱饵蛋白(prey)与NubG融合,猎物蛋白(bait)与Cub-LexA-VP16融合。,当当诱饵和猎物发生相互作用时, NubG 和 Cub 的相互接近,结合,形成完整泛素ubiquitin,从而被 UBPs 酶识别,导致 LexA-VP16 的剪切,进入核内,从而激活报告基因AUR1-C、ADE2、HIS3和MEL1的转录。
① 酵母杂交文库构建
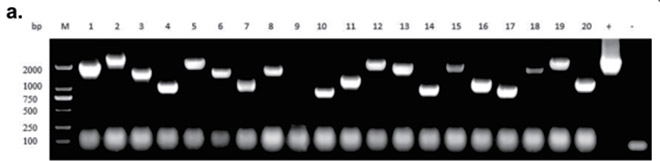
图⽚来源于 Xu et al. BMC Biotechnology (2020) 20:4
② 酵母双杂交筛选
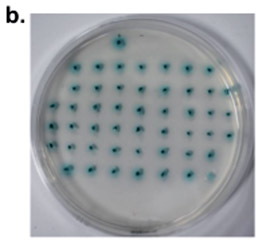
阳性结果点钟SD/-Leu/-Trp/-HIS3/-ADE2+X-α-gal平板
图⽚来源于 Xu et al. BMC Biotechnology (2020) 20:4
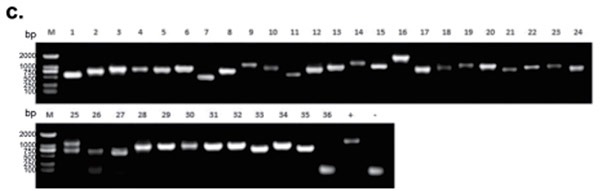
阳性结果PCR鉴定
图⽚来源于 Xu et al. BMC Biotechnology (2020) 20:4
③ 酵母杂交验证(可用于文章发表)
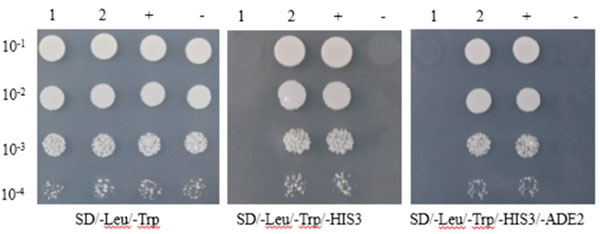
1: PGBKT7-Bait+PGADT7(⾃激活验证)
2: PGBKT7-Bait+PGADT7-Prey(实验组)
+: PGBKT7-53+PGADT7-T(阳性对照)
- : PGBKT7-lam+PGADT7-T(阴性对照)
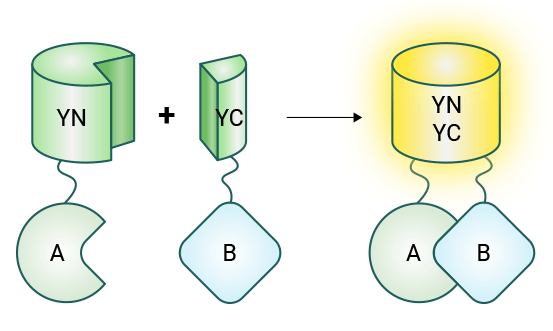
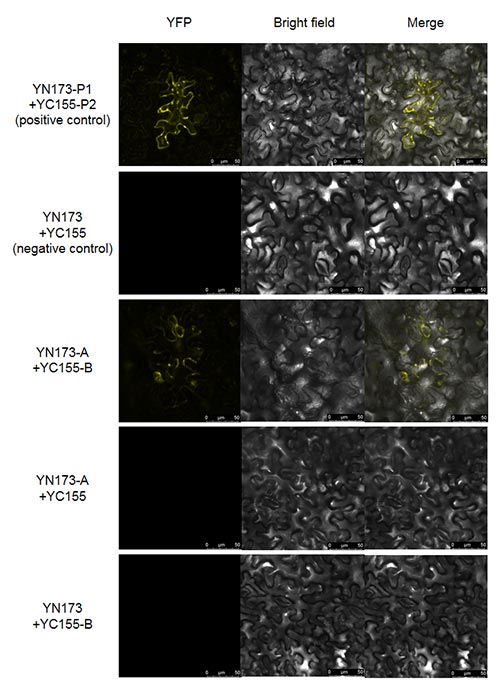
双分子荧光互补(Bimolecular Fluorescent Complimentary,BiFC)技术基于蛋白质片段互补的原理,用于研究蛋白质间的相互作用。其原理是将荧光蛋白(如绿色荧光蛋白GFP、黄色荧光蛋白YFP等)切割为两个互补但各自不发光的片段(如N端和C端片段),分别与待检测的蛋白质A和B融合表达。当蛋白质A与蛋白质B在细胞内发生相互作用时,荧光蛋白片段靠近并重组为完整的荧光蛋白,从而在显微镜下产生荧光信号,直观地反映了蛋白质间的相互作用。
分子、细胞、个体水平上的机制研究、功能研究、表型研究是生物学研究中的热点。分子(核酸与核酸、核酸与蛋白、蛋白与蛋白)互作研究是机制研究的重要组成部分,是功能、表型研究的进一步深化。随着人类基因组计划的完成,后基因组时代的深入,测序、质谱、生物信息联合分析等技术的进一步发展,使得高通量筛选生物标记物、寻找生物关联分子变得可能,同时对分子互作技术的应用提出了更高的要求。
金开瑞多年来致力于分子互作机制研究,组建了专门的分子互作研究技术团队,拥有丰富的互作类项目设计、实施经验,可以为您提供核酸与核酸、核酸与蛋白、蛋白与蛋白间等各类从方案流程设计到技术实施的整套互作研究服务。
此外,金开瑞生物还特别推出了多款高效、简便且经济实惠的互作分析小规格试剂盒,包括但不限于RIP kit(6T)、Co-IP kit(动物/植物)(6T)、ChIP kit(动物/植物)(6T)、RNA pull-down kit(6T)、银染试剂盒(10T/20T/40T)、DNA pull-down kit(动物/植物)(6T)和双荧光素酶报告基因检测试剂盒(50T),旨在为您的研究提供更多的选择,欢迎采购。











