HI-C技术(三维基因组)经典文献解读
信息来源:金开瑞 作者:genecreate 发布时间:2018-10-24 17:29:11
题目:Genome organization and chromatin analysis identify transcriptional downregulation of insulin-like growth factor signaling as a hallmark of aging in developing B cells
通过基因组组织和染色质结构分析确定了胰岛素样生长因子信号的转录下调是B细胞开始衰老的标志
期刊:Genome Biology
影响因子:13.214
研究背景
衰老的特征是适应性免疫系统功能的丧失,但其根本原因却鲜为人知。为了评估年龄对B细胞发育的影响,作者对组蛋白修饰和染色体构象相关的基因表达和染色质特征进行了分析,使用的实验材料来自不同年龄阶段的小鼠的pro-B和pre-B细胞。
研究内容及结果
1. 在衰老过程中,平均pro-B和 pre-B细胞数量分析
使用多参数流式细胞分析小鼠骨髓B细胞,发现老年小鼠的pre-B细胞发生实质性的变化(图1),老年的(三个月)与年轻的老鼠(约19 - 22日)相比,pro-B细胞大约减少了两个(图1 c)、pre-B细胞却有着近两倍数量的减少(图1 d),该结果与先前的报道一致。与此相反的是,发现在老年小鼠骨髓中会重新循环成熟的B细胞(图1e),不能排除被分类的衰老pro-B细胞群体中存在少量成熟B细胞污染。可能是由于成熟B细胞再循环数量增加,以及在衰老的骨髓中,B细胞前体(IgM-)和成熟细胞(IgM+)分离不那么明显。
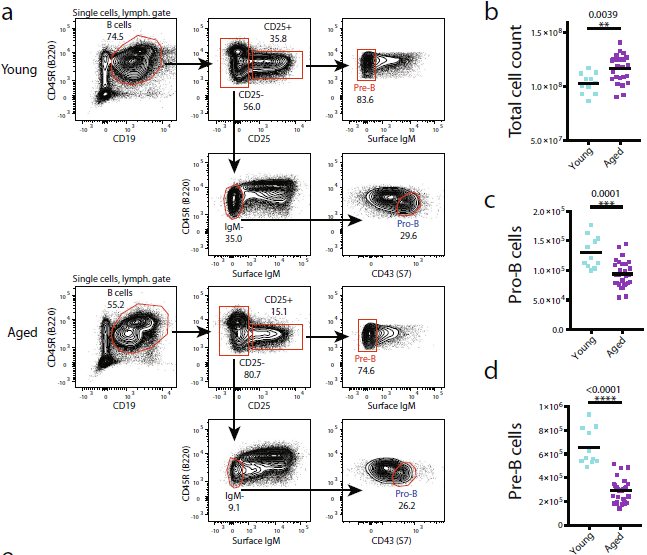
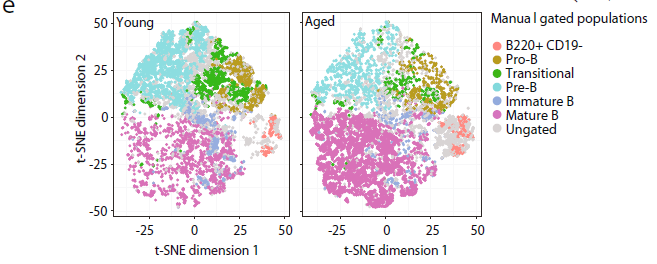
图1 衰老小鼠B细胞前体基因表达变化
2. pro-B和pre-B 细胞中的基因表达分析
对核糖体RNA (rRNA)耗尽的总RNA转录组分析发现,衰老pro-B细胞中有82个显著上调和54个下调基因,衰老pre-B细胞中有23个上调和33个下调基因(图2 a , b)。pro-B细胞和pre-B细胞之间的年龄特异性表达变化是相关的,即使只有一种细胞类型也能达到显著性(图2 c)。在pro-B和pre-B细胞阶段的老化过程中,一些基因被降低了,这些基因编码胰岛素样生长因子(IGF)信号传导途径的组成部分,如IRS1和IGF1r。IRS1是通路的一个关键组成部分,被发现其是衰老pre-B细胞信使RNA (mRNA)水平上最显著的下调基因。因此,作者研究了IRS1蛋白水平,发现在pro-B细胞和pre-B细胞的老化过程中,蛋白含量都有所下降,这表明IRS1介导的特定年龄mRNA变化会降低蛋白质水平(图2d)。对于衰老前B细胞中下调的基因,在32个启动子中,有19个启动子的活性启动子或活性调控区域状态被降低。与大多数上调基因的观察结果相似,这些变化大多是细微的,与这些基因表达中相对较小的折叠变化相一致(图2b)。
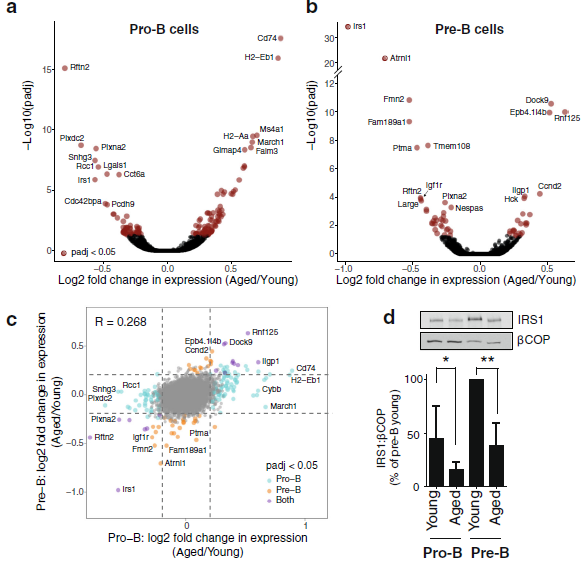
图2 基因表达在pro-B和pre-B 细胞随着年龄的老化而改变
3. 初期的pro-B 和pre-B细胞的RNA和microrna表达分析
总RNA水平既反映了转录速率,也反映了RNA稳定性等下游过程。为了更直接地探讨衰老对基因转录的影响,作者从年轻和衰老的pro-B细胞中分离出核RNA,以丰富新生转录本,并以内含子转录的整体变化作为新生转录的一种具体衡量指标。同时,在总RNA中检测到17%(175个中有30个)的DEGs在核RNA测序(RNA-seq)分析中也是DEGs,并且整体的折叠变化是相关的,从而可以认为更多的基因存在与年龄相关的显著性差异转录(图3a)。值得注意的是,一些年龄下调基因,如Plxdc2、Igf1、Igf2bp3和Igf1r,以及上调的基因,如Adam19和Tmem163,都与IGF信号或2型糖尿病有关。更广泛地说,DEGs的KEGG 通路分析突出了几个与营养信号相关的代谢通路。
通过核RNA-seq分析显示Nespas是衰老pro-B细胞中最显著的下调基因之一(图3a)。Nespas是非编码转录本,参与调控印迹,是miRNAs miR-296和miR-298的非编码前体RNA。为了探索B细胞前体中miRNA表达与衰老的关系,作者进行了小RNA-seq,在pro-B细胞和pre-B细胞中鉴定出34个有差异显著的mirna(图3b、c)。其中20.6%在pro-B细胞和pre-B细胞中均表现出表达差异。该分析证实了miR-296和miR-298的显著下调,与新生RNA-seq检测到的Nespas水平变化一致。作者还观察到7个Let-7家族基因和miR-223在pro-B和pre-B细胞衰老时上调(图3b, c)。不同表达的microrna在衰老过程中被分离成簇,表达变化相似,并且其中一些表达在pro-B细胞和pre-B细胞之间也有调节作用(图3c)。
Let-7 mirna由12个具有相同序列的成员组成,分别从基因组位点Let-7a1、Let-7a2、Let-7b、Let -7c、Let -7c、Let-7d、Let-7e、Let-7f1、Let-7f2、Let-7g、Let-7i和miR-98进行表达。Let-7b和7 c在年老的pre-B细胞显著上调microrna,作者检查了核RNA-seq数据,年老的pro-B细胞非编码RNA 在15号染色体上显著增加。因此推测,这个转录组可能是这些microrna的RNA前体。
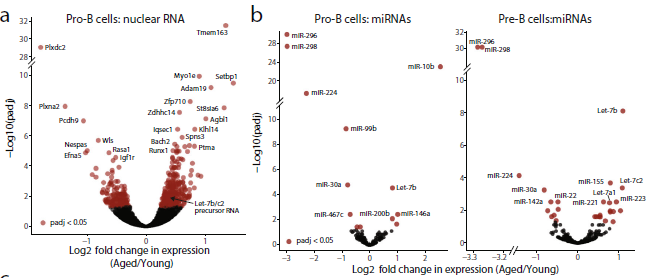
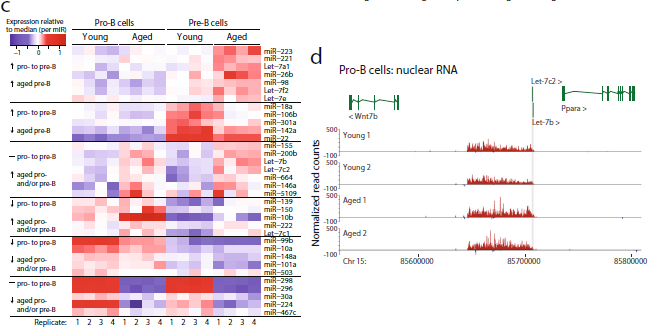
图3 初期的pro-B和pre-B细胞的RNA和microrna表达随着年龄的老化而改变
4. 染色质可及性和pre-B细胞中的H3K27me3与年龄相关的变化分析
在老年pre-B细胞中,发现10个可及性明显较低的区域和4个可及性较高的区域(图4a)。可及性低的两个区域与Irs1启动子区域重叠,另一个区域与Irs1转录终止位点接近(图4a);对于组蛋白修饰物H3K27me3,只确定了两个峰,在年轻和衰老pre-B细胞之间存在显著的富集差异(图4b),第一个峰位于Irs1启动子上,在衰老pre-B细胞中表现出显著的富集,与Irs1转录下调一致,第2个峰位于15号染色体上Let-7b/c2调控区域(图4b)。
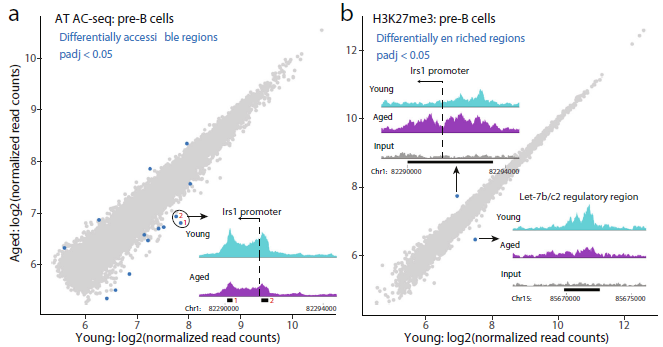
图4 染色质可及性和pre-B细胞中H3K27me3与年龄相关的变化分析
5. 基因表达的变化与老化的B细胞前体染色质变化分析
如图5所示,基于阈值方法识别高置信度位点,显示出染色质可及性的变化和组蛋白在衰老过程中的修饰变化非常少,仅显示了Irs1和Let-7 mirna转录调控的重要染色质成分。这表明染色质随着年龄变化而的变化,具体体现在H3K4me3和H3K27me3的占有率,这一现象可能对基因表达有影响,但进一步研究发现,如果有基因表达发生了变化,也会引起相关的染色质发生变化。值得注意的是,H3K4me3和H3K27ac更符合差异基因表达而不是染色质可及性。
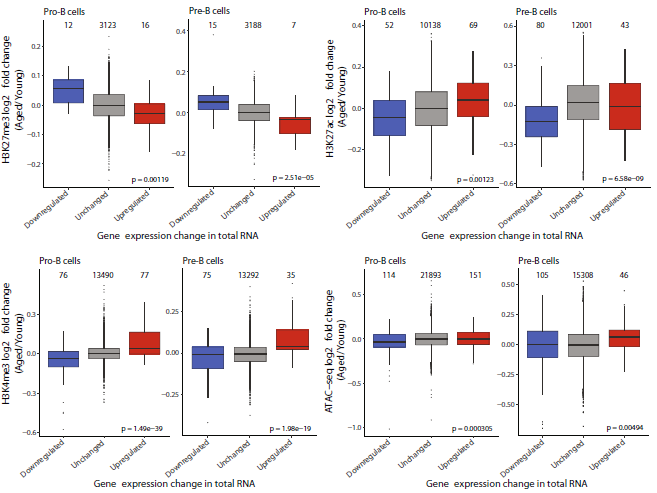
图5 基因表达的变化与老化的B细胞前体染色质变化
6. 在pre-B细胞中进行染色质状态分析,识别与年龄相关的变化
基于平衡类内同质性和类间异质性,作者选择了一个的16状态模型(图6a)。该模型分为6大类,称为“多梳”、“双价”、“活性启动子”、“活跃的调节区域”、“绝缘体”和“背景”。为了整合这些变化,并确定在染色质状态下启动子表现出类似转变的基因组,作者使用无聚类法。将启动器分隔为九个显示不同状态变化模式的集群,这些基因也表现出与染色质状态改变相一致的基因表达调控机制。例如,从主动启动子状态到双价或多梳状态的基因表达,通常在衰老的时候会减少。相反,显示相反转变的基因(聚类6)或其多梳状态占用减少的基因(聚类5)倾向于表达增加(图6 c)。
作者接下来评估了所有基因启动子的染色质状态变化,这些基因在pre-B细胞中表现出明显的年龄特异性表达变化。年龄上调基因的启动子只出现了细微的变化,也有少数例外,包括新发现的Let-7b/c2前体基因、Ccnd2 (cyclin D2)和Mctp1。启动子区域从年轻时的二价显性状态转变为年老的pre-B细胞更活跃状态(图6d)。在全基因组分析中,Ccnd2和Mctp1都被分配到6组(图6c)。另外观察到Irs1启动子上发生了实质性的变化,随着年龄的增长,活跃状态转变为二价状态(图6d),在全基因组分析中也发现了这一点,在集群2中分离(图6c)。因此,染色质状态分析显示了在启动子上染色质改变的基因。
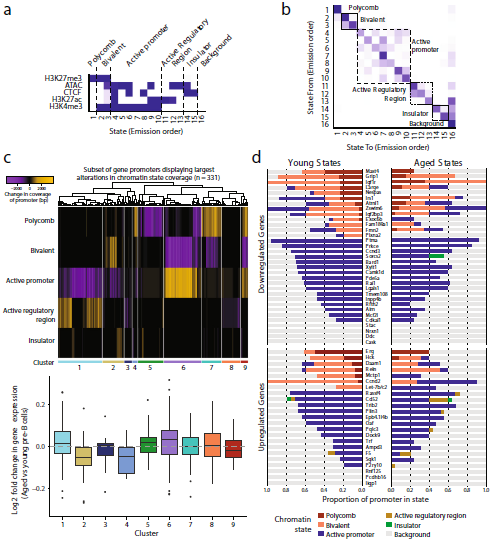
图6 pre-B细胞中染色质状态分析
7. pre-B细胞的基因组组织变化
衰老与基因组组织的变化有关,为了研究B细胞发育过程中是否会发生这种变化,以及是否与基因调控的变化相关,作者首先在B细胞前期分离的细胞核中进行了Hi-C检测。利用主成分分析(PCA)和H3K4me3数据,使用Hi-C数据将基因组分为A(活跃的)和B(压抑的)两部分。这一分析表明,年轻和老年pre-B细胞之间的染色体间隔几乎相同。在9928个区域中,有100个区域在老化后的隔间得分有显著变化(图7a,左上),40个区域的隔间得分显著增加,其中3个区域从压抑的B隔间(负分)变为活跃的A隔间(正分)。相反,60个区域的间隔分数显著降低,有6个从A隔间切换到B隔间。值得注意的是,A-to-B转换区域之一包含1号染色体上的Irs1基因(图7a,下),间隔评分降低的区域与10个基因重叠,这些基因在衰老pre-B细胞中表达显著降低,其中包括Plxna2和Igf1r。另外,发现随着隔间分数的增加基因表达呈高度显著的增加趋势,反之亦然(图7a,右上方)。这表明,在活跃和压抑的核环境之间基因的重新定位可能有助于在衰老过程中调节基因表达。
为了获得pre-B细胞前期启动子相互作用及其衰老动力学的高分辨率图像,作者进行了启动子捕获Hi-C(PCHi-C),对年轻和老年小鼠的细胞进行研究,并利用CHiCAGO试验确定具有统计学意义的启动子相互作用。为了区分这种相互作用,作者分析了启动子PIR相互作用。与其他细胞类型的PCHi-C结果一致,发现PIRs富集于与基因调控相关的特征,包括H3K4me3、H3K27me3和H3K27ac(图7b)。这表明,许多检测到的物理交互作用将启动子与调控元件连接起来。上调基因的启动子在衰老pre-B细胞中获得了与其他启动子的相互作用,并且很少与启动子或PIRs失去相互作用,而下调基因在衰老过程中经常与两者失去相互作用(图7c)。随后分析CHiCAGO互动分数的变化,这显示了上调基因分数的普遍趋势,存在更强或更频繁的相互作用,而下调基因的得分较低,提示与调控元件失去联系(图7d)。总的来说,作者没有发现基因表达变化与衰老后PIRs染色质改变之间有显著的联系(图7e),一些被下调的基因失去了与活跃区域的相互作用,这一趋势没有达到显著性(图7f)。
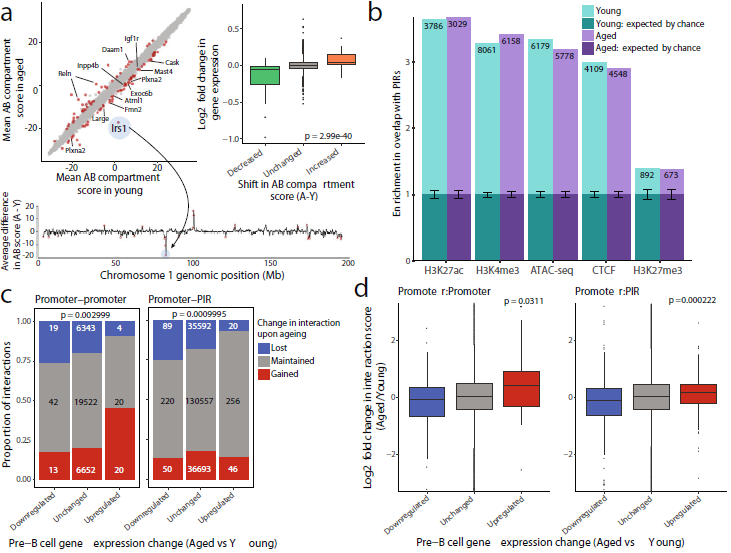
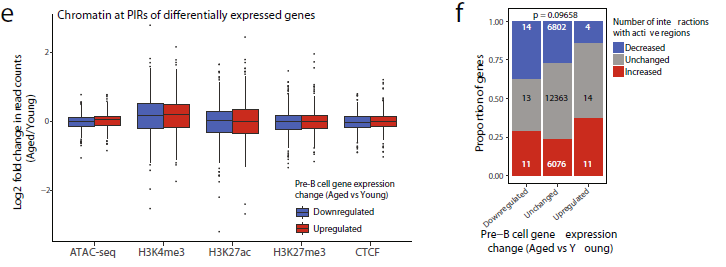
图7 B细胞前体基因组组织的年龄特异性变化
8. 衰老pre-B细胞启动子相互作用的重新连接分析
作者接下来探讨了不同年龄的基因表达变化与PIRs染色质状态变化之间的关系。在某些情况下,例如在P2ry10、Epb4.1l4b和Ccnd3基因上,上调或下调基因的PIR分别显示活跃染色质标记的水平略有增加或减少(图8b)。作者还观察了一些上调基因启动子与染色质活跃区(包括启动子和PIRs)连接的案例,值得注意的是,这种相互作用的“重新连接”的例子包括随年龄下降的Irs1基因,它在衰老细胞中丢失了一些相互作用,例如与Cul3启动子周围高度活跃的染色质的相互作用(图8a)。这些结果表明,在衰老过程中,即使启动子的表达发生了改变,染色质在PIRs上的调节并不经常发生。随着年龄的增长,基因表达的变化通常与特定启动子相互作用的“重新连接”有关。这可能有助于在活跃(A)和非活跃(B)基因组环境之间进行更广泛的转换。
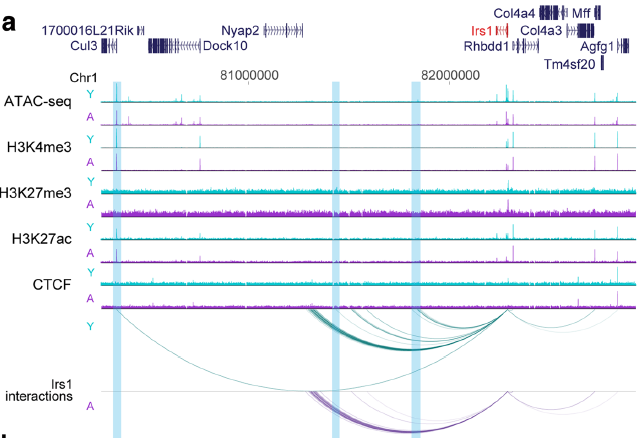
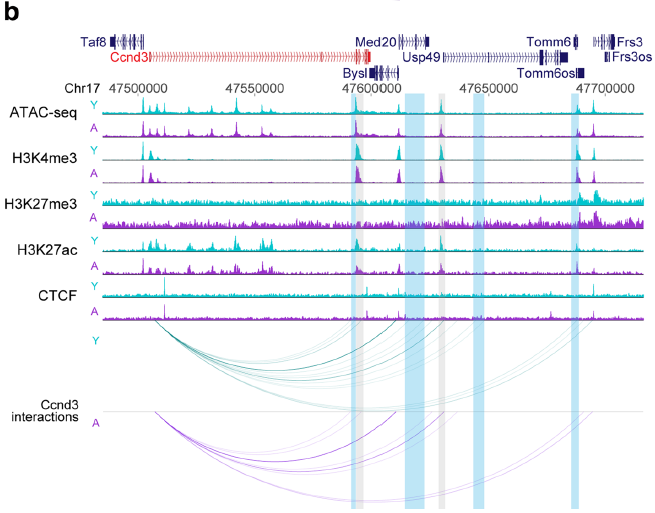
图8衰老pre-B细胞启动子相互作用的重新连接
文章小结
与幼龄鼠相比,老年小鼠的大多数基因的表达水平通常被保存在pre-B细胞中。但是,许多基因表达会有年龄特异性的变化,包括microRNA编码基因。这些变化是由染色质结构的多层改变所引起的,包括染色质的可及性、组蛋白修饰、远程启动子互动和核区划分。先前的研究表明,年龄差异化与促进调节元素交互的变化有关。作者发现,在衰老B细胞前体上,会重新连接原有的差异化与促进调节元素相互作用。最终,作者确定了胰岛素样生长因子(IGF)信号通路的转录负调控机制,特别是对Irs1和Let-7 microRNA表达的负转录调控,这是一项重要的老年表型特征。这些变化与H3K27me3的具体改变有关,这表明,多细胞介导的抑制在pre-B老化中起着重要作用。
解析文献
Hashem Koohy, Daniel J, et al. Genome organization and chromatin analysis identify transcriptional downregulation of insulin-like growth factor signaling as a hallmark of aging in developing B cells. Genome Biology , 2018 , 19 (1) :126
参考文献
1. Imakaev M, Fudenberg G, et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nat Meth. 2012;9:999–1003.
2. Wingett S, Ewels P, et al. HiCUP: pipeline for mapping and processing Hi-C data. F1000Res. 2015;4:1310.
3. Mifsud B, Tavares-Cadete F, et al. Mapping long-range promoter contacts in human cells with highresolution capture Hi-C. Nat Genet. 2015;47:598–606.
4. Cairns J, Freire-Pritchett P, et al. CHiCAGO: robust detection of DNA looping interactions in Capture Hi-C data. Genome Biol. 2016;17:127.
最新动态
-
04.22
一文读懂EMSA技术核心要点,让“emsa” 秒变“easy”
-
04.02
4·2世界孤独症日 | 聆听“星”声“泌”语,让爱来,让碍走
-
04.01
酵母杂交核心技术:深度Q&A帮你轻松突破实验瓶颈
-
03.19
【客户文献解读,IF>11】食管癌的"隐形推手":MALR-ILF3-HIF1a轴的强大作用
-
03.18
siRNA介绍及药物研发的现状前景
-
03.18
知无不“研”|5篇高分文献带你一览高通量酵母杂交的非凡魅力~
-
02.27
【客户文章分享】SHMT2 通过 5′UTR 依赖性 ADAM10 翻译启动介导小分子诱导的阿尔茨海默病病理学缓解过程
-
01.24
客户文献解读 | 中医为什么能治流感?是玄学还是运气?INT J NANOMED揭示鱼腥草抗病毒机制及范围!
-
01.24
客户文献分享,IF>11|Shank3:脑缺血再灌注损伤的守护者,揭示神经保护的新篇章
-
12.27
文献解读 | 高密度脂蛋白通过miR-181a-5p调控自噬影响血管新生










