RNA蛋白免疫沉淀的原理(RIP实验)
RNA蛋白免疫沉淀(RNA-protein immunoprecipitation,简称RIP)是常用的实验技术,用于研究RNA与特定蛋白之间的相互作用。其原理基于抗体的特异性结合,使目标RNA与与之相互作用的蛋白结合,并随后通过免疫沉淀分离出这些RNA蛋白复合物。
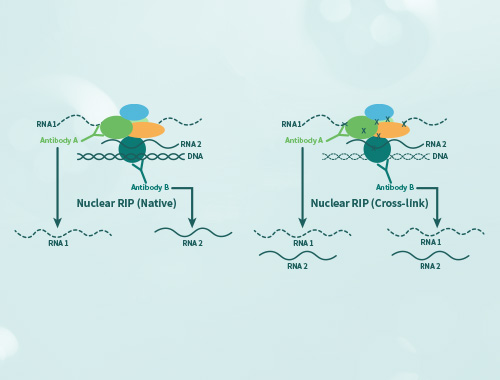
以下是RIP实验的一般步骤和原理:
交联:将细胞或组织中的RNA与与之相互作用的蛋白交联在一起,通过化学交联剂(如甲醛)或者紫外线辐射进行交联。
细胞裂解:将交联的细胞或组织样品裂解开,释放出细胞内的RNA蛋白复合物。
免疫沉淀:将目标蛋白的特异性抗体加入至裂解液中,利用抗体与蛋白的特异性结合,将目标蛋白及其相关的RNA结合成复合物。
洗涤:通过多次洗涤步骤,去除非特异性结合的蛋白和RNA。
分离:将RNA蛋白复合物从剩余的细胞裂解产物中分离出来,通常采用磁珠等辅助材料。
去交联:将RNA蛋白复合物去除交联,通过加热或酶消化等方法解除RNA与蛋白间的共价键。
RNA提取:从复合物中提取RNA,以供后续实验分析,如逆转录聚合酶链反应(RT-PCR)或高通量测序等。
通过RIP实验,可以确定特定蛋白与RNA之间的相互作用,并进一步了解这些作用在细胞中的功能和调控机制。
最新动态
-
01.23
面向临床转化的外泌体研究方案,如何建立体液外泌体无创检测体系,实现疾病的动态监测、疗效评价与复发预警?
-
01.23
细胞凋亡和细胞坏死的区别是什么?
-
01.20
重组蛋白的制备流程是什么?
-
01.20
rna原位杂交的主要实验过程及应用有哪些?
-
01.20
多克隆抗体的优势和局限性分别体现在哪些方面,适合应用于哪些研究场景?
-
01.19
蛋白和蛋白相互作用怎么验证?
-
01.19
双荧光素酶报告基因实验用于评估药物筛选与机制解析
-
01.19
单克隆抗体制备为什么不用浆细胞检测?
-
01.19
噬菌体筛选文库构建时双峰较多会影响后续筛选吗
-
01.16
EMSA凝胶电泳为什么必须使用非变性聚丙烯酰胺凝胶?










