Nature Genetics丨三维基因组学研究新方法获重要进展
信息来源:金开瑞 作者:genecreate 发布时间:2018-05-02 17:01:41
4月26日,武汉金开瑞生物工程有限公司技术顾问、华中农业大学教授, 曹罡课题组与华中农业大学李国亮教授课题组在国际著名期刊Nature Genetics上联合发表题为“Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture”的研究论文。该论文主要介绍了一种新的染色体构象捕获技术(DLO Hi-C),为解析基因组三维结构提供了一种新型、高效、经济的研究方法。

基因组三维空间结构与功能的研究简称三维基因组学(Three-Dimensional Genomics,3D Genomics),是指在考虑基因组序列、基因结构及其调控元件的同时,对基因组序列在细胞核内的三维空间结构,及其在基因转录、调控、复制和修复等生物过程中功能的研究。染色体是由DNA与组蛋白共同组成,从染色体的一级结构(绳珠模型)到四级超螺旋折叠结构,DNA分子一共被压缩了8400倍左右,正是这些折叠和压缩,导致基因在细胞中的分布复杂而又有序,只有了解清楚染色体区域(A/B compartments、TADs、Loops),才可以将基因组上原本分散的远距离调控元件与其具体调控区域更好的关联起来,对理解基因的转录调控、增强子与启动子的相互作用、疾病易感位点、DNA损伤修复、基因组结构变异和表观遗传有着重要的意义。
科学家们对染色体三维结构的研究很早就开始了,但是相关的研究一直不够深入,同时缺乏微观的证据。直到2003年Job Dekker及其合作者提出了染色质构象捕获技术(Chromatin Conformation Capture,3C),用于测定特定的点到点之间的染色质交互作用。随后,科学家们扩展了3C技术,开发了4C技术(Circularized Chromatin Conformation Capture),用于测定一点到多点之间的染色质交互作用。Dostie等人接着开发了5C技术(Carbon-Copy Chromatin Conformation Capture),用于测定多点到多点之间的染色质交互作用。
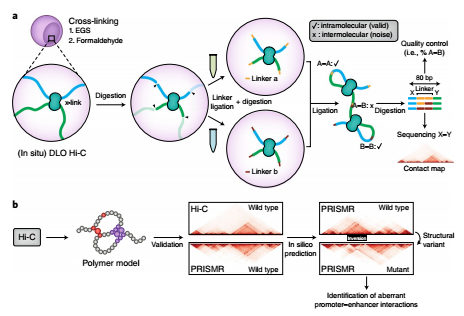
为了能捕获全基因组范围的染色质相互作用,Job Dekker研究组又开发出了现在大家所熟知的Hi-C技术。Hi-C技术是三维基因组学的主要研究方法,但存在实验成本高、数据噪音大、实验过程繁琐等缺点。为解决这些问题,曹罡教授及其合作者提供了一种新的DLO Hi-C染色体构象捕获技术(digestion-ligation-only Hi-C , DLO Hi-C),设计了巧妙的酶切位点,采用同时酶切酶连的方式,将DNA接头连接在染色体内切酶切口末端上,然后进行邻近酶连,最后再用MmeI内切酶酶切消化,回收固定大小互作DNA片段,从而大大地降低了测序背景数据噪音,获取的测序数据质量高于传统Hi-C。DLO Hi-C技术还可以用于染色体易位位点筛选。
科学家们对染色体三维结构的研究很早就开始了,但是相关的研究一直不够深入,同时缺乏微观的证据。直到2003年Job Dekker及其合作者提出了染色质构象捕获技术(Chromatin Conformation Capture,3C),用于测定特定的点到点之间的染色质交互作用。随后,科学家们扩展了3C技术,开发了4C技术(Circularized Chromatin Conformation Capture),用于测定一点到多点之间的染色质交互作用。Dostie等人接着开发了5C技术(Carbon-Copy Chromatin Conformation Capture),用于测定多点到多点之间的染色质交互作用。
为了能捕获全基因组范围的染色质相互作用,Job Dekker研究组又开发出了现在大家所熟知的Hi-C技术。Hi-C技术是三维基因组学的主要研究方法,但存在实验成本高、数据噪音大、实验过程繁琐等缺点。为解决这些问题,曹罡教授及其合作者提供了一种新的DLO Hi-C染色体构象捕获技术(digestion-ligation-only Hi-C , DLO Hi-C),设计了巧妙的酶切位点,采用同时酶切酶连的方式,将DNA接头连接在染色体内切酶切口末端上,然后进行邻近酶连,最后再用MmeI内切酶酶切消化,回收固定大小互作DNA片段,从而大大地降低了测序背景数据噪音,获取的测序数据质量高于传统Hi-C。DLO Hi-C技术还可以用于染色体易位位点筛选。
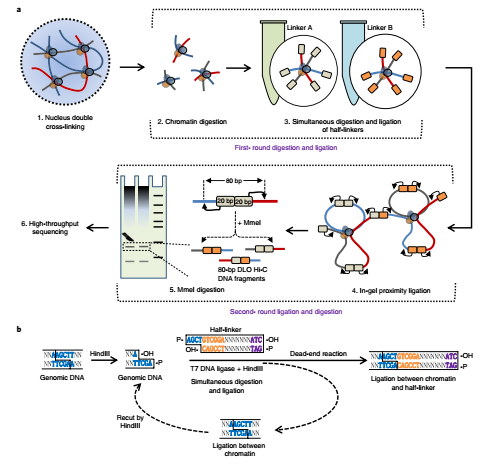
DLO Hi-C技术使全基因组染色体构象捕获实验的成本大大的降低,同时简化了实验步骤,使得实验成功率显著提高,对辅助基因组组装、解析基因组远程调控元件的功能、理解疾病易感位点以及检测染色体结构变异有着重要的意义。Nature Genetics同期发表专门评论性文章,对曹罡教授及其合作者的研究工作给予了较高评价“Lin et al. introduce an elegant dual-linkerstrategy that allows for noise filtering and early quality control.”
随后还将详细解读该篇论文,从实验设计、流程、生信分析及可视化方面进行详细说明,干货满满,敬情期待哦!
文章链接:https://www.nature.com/articles/s41588-018-0111-2
文章链接:https://www.nature.com/articles/s41588-018-0111-2
最新动态
-
01.16
新源地,共华章,开瑞景 ——武汉生之源生物科技股份有限公司暨子公司乔迁庆典圆满落幕
-
12.10
【直播】酵母双杂交技术:从原理逻辑到案例应用的系统精讲
-
10.24
【直播】荧光技术深度解析-双荧光素酶、BIFC、LCA的应用与选择
-
09.18
【直播】分子生物学核心技术详解
-
08.14
【直播】GST pull-down原理、应用与优化实战
-
07.24
【直播】解密基因调控:ChIP技术全攻略
-
06.19
【直播】免疫共沉淀(CoIP)实验流程和常见问题解决
-
05.15
【直播】EMSA技术全解析-助你攻克实验难点
-
05.14
2024年金开瑞危险废物污染环境防治信息公开
-
04.16
【直播】解码动物外泌体:从基础科研到转化应用突破










