一文带你全方位解析ChIP技术
染色质免疫沉淀(Chromatin Immunoprecipitation, ChIP) 作为解析DNA-蛋白质相互作用的经典方法,自1984年由Gilmour和Lis首次建立以来,已成为表观遗传学研究的关键技术。该技术通过抗原-抗体特异性结合原理,实现对目标蛋白(如转录因子、修饰组蛋白)及其结合基因组DNA片段的共沉淀,从而精确定位蛋白-DNA互作位点。在基因表达调控、表观遗传修饰图谱构建及疾病发生机制研究中,ChIP技术提供了不可替代的空间分辨率证据,为揭示真核生物转录调控网络奠定了方法论基础。
一、技术原理:ChIP如何“锁定”目标?
我们可以把ChIP想象成为一场精密的“分子抓捕行动”:
(1)交联:甲醛快速“冻结”细胞内DNA与蛋白质的相互作用;
(2)片段化:超声波或酶将染色质“粉碎”成200-500bp小片段;
(3)免疫沉淀:特异性抗体如“磁铁”般吸附目标蛋白及其结合的DNA片段;
(4)解交联与纯化:释放DNA片段并纯化;
(5)下游分析:qPCR或测序揭示蛋白质结合的DNA序列。
(6)整个过程如同在浩瀚基因组中精准定位目标蛋白的“专属车位”!
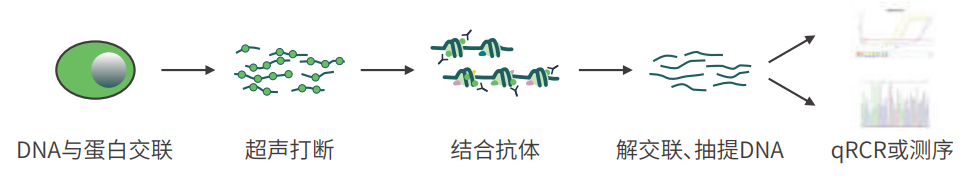
图1. ChIP实验流程
二、主要应用场景
1、转录调控
锁定转录因子(如p53, NF-κB)在启动子、增强子上的结合位点,揭示基因开关机制。
例如:发现癌基因MYC的关键调控位点,助力靶向药物设计。
2、组蛋白修饰
检测组蛋白修饰(H3K4me3激活标记,H3K27me3抑制标记),绘制表观遗传图谱。
应用:干细胞多能性维持、细胞命运转变研究。
3、疾病机制
追踪疾病中异常蛋白-DNA互作(如肿瘤中BRCA1结合异常),发现治疗新靶点。
突破:揭示阿尔茨海默病中组蛋白修饰紊乱。
三、如何选择:ChIP-qPCR vs ChIP-seq
当需要解析蛋白质与DNA的互作关系时,这两个技术该如何选择呢?我们先来看下两个实验的区别在哪里!
|
特性 |
ChIP-qPCR | ChIP-seq |
|
检测范围 |
已知特定靶位点(如启动子) | 全基因组无偏扫描 |
|
分辨率 |
单个位点精确定量 | 全基因组高分辨率图谱 |
|
通量 |
低(单次几个位点) | 高(一次覆盖全基因组) |
|
成本 |
低(无需测序) | 高(依赖高通量测序) |
|
适用阶段 |
验证候选区域/预实验优化 | 探索未知互作区域/全局分析 |
选择策略:
✅ 目标明确(如验证TF结合已知启动子)→ ChIP-qPCR
✅ 探索未知区域/绘制全基因组图谱 → ChIP-seq
四、实验成败关键点!
样本制备:质量是试验成功与否的重要前提。
动物组织:新鲜组织需要用PBS洗涤去除残留血液和污染物,迅速剥去脂肪和筋皮等结缔组织,冲洗干净;用组织剪或手术刀将组织分离成1cm3左右的小块,装入2mL或者更大体积的螺旋冻存管中,准确标记名称。
植物组织:如果样品表面有泥土或明显污染物,先用PBS清洗,吸水纸吸去表面液体。
样本量:哺乳动物细胞≥5×106(注意:活率85%以上),组织根据实际情况进行样本准备,柔软组织3-5g即可,坚硬组织建议10g以上。
交联时间:1%甲醛,室温10-15分钟(过长导致抗原遮蔽!)
终止:甘氨酸至终浓度0.125M
超声破碎:均匀片段化是核心,参考条件:18%或50W、ON 1S、OFF 1S,超声15min
目标:获得合适得染色质片段,ChIP-qPCR片段集中在200-700 bp,ChIP-seq片段集中在300bp左右最佳
抗体选择:选错抗体会导致实验失败,必选经ChIP验证的抗体(查看文献/官网数据),因为它能高特异性识别目的蛋白的立体表位,且在通过IP和洗涤步骤后仍然能够保持与目的蛋白的紧密结合。建议开展预实验(如Western或IP)验证抗体有效性
避免陷阱:
➤仅WB验证的抗体可能无效!
➤多克隆抗体需警惕批次差异
五、进阶攻略:多组学联合揭示全局调控
当研究基因表达调控网络时,单一技术仅能捕捉分子事件的单一维度:
ATAC-seq:鉴定染色质开放区域(潜在调控元件),但无法明确具体作用蛋白
ChIP-seq:定位特定蛋白结合位点,但无法区分功能性结合与非功能性结合
我们可以让ChIP+ATAC-seq强强联合,解锁更高维调控逻辑!
ATAC-seq:先快速扫描全基因组开放染色质区域(潜在调控元件)
ChIP精准定位:在开放区域锁定特定TF或组蛋白修饰
整合分析:
① 重叠ChIP峰与ATAC峰→验证功能性结合位点
② 关联RNA-seq数据→建立“染色质开放-蛋白结合-基因表达”因果链
六、常见问答
1.抗体的需要用量?需要准备多少抗体?
在ChIP实验中,一个免疫沉淀反应使用5μg左右的抗体量,具体用量需要通过摸索调试,若效价不佳可能需增加到10μg左右,因此做一个样本需要准备的抗体量至少应>10μg或10μL。
2.染色质片段大小在哪个范围比较合适?
对于ChIP-seq,片段在200-500bp左右是最合适范围;对于ChIP-qPCR,片段在200-800 bp左右适宜。
3.ChIP实验植物样本处理和动物组织/细胞有何区别?
植物组织由于细胞壁、气腔等结构的存在,会给交联缓冲液的作用带来困难,因此相对于动物组织/细胞来说,往往需要在抽真空条件下进行交联,而该步骤是一个需要经验及优化的过程。
4.ChIP实验如何设置对照及重复?
通常ChIP过程产生3份DNA产物,Input、IgG产物、IP产物。Input是染色质片段化后提取获得的总DNA、未经抗体加入免疫沉淀过程,IgG组这是用实验抗体的同型阴性对照抗体、作为mock IP,IP产物即是以实验抗体获得的免疫沉淀产物。
在后续的qPCR检测中,通常将IgG产物组作为IP产物组的对照。如需设置阳性对照抗体,可以选择Histone H3或者RNA polymerase II,然后以GAPDH或Actin等作为检测基因。
在后续的Sequence中,通常将Input组产物作为测序对照样品。
5.我们ChIP-qPCR中对于一个基因启动子设计多少引物?
如果未能确定启动子上的具体结合位点,则需要对启动子的-2k范围设计qPCR引物,建议至少设计3对引物,有时存在特殊性的启动子可能会增加到4对引物。引物设计过程是:找到已经研究确认的被调控基因、或本实验中欲验证的基因,选择其启动子区域(一般从转录起始位点上溯到-2k),以该序列为模板设计qPCR引物,如研究的转录因子具有特定的结合序列motif,则可着重在这些motif附近设计引物。
6.ChIP-Seq中的测序DNA样本需要多少产量?
通常是≥10ng。随着目前建库技术的不断完善,低至5ng的DNA量也能被建库成功;少数测序公司为了降低自身风险提出20ng的要求,通过可以协商降低该送样要求。尤其是一些转录因子,最终能够捕获的DNA量会很低。
7.我所研究的蛋白难以找到ChIP级抗体怎么办?
较常用的方式可以将目的蛋白同某种标签融合表达,比如HA、GFP、Myc等标签,使用这些标签抗体的ChIP级抗体,这种途径也常见于发表文献中。
8.ChIP风险如何判断?
ChIP实验以标签来判断实验风险,重组标签的转录因子>内源转录因子>组蛋白;当以重组蛋白作为靶蛋白时,重组蛋白同内源蛋白可能存在结合活性、结合位点差异;以标签抗体进行ChIP时、染色质结合位点本身会被内源蛋白竞争,这些都会影响到ChIP过程的特异性捕获效率。
最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










