RIP-qpcr蛋白分析实验步骤
RIP-qPCR是一种用于研究RNA结合蛋白与靶RNA相互作用的实验方法。本文将详细介绍RIP-qPCR的实验步骤。实验步骤如下:
1、细胞裂解
将需要研究的细胞或组织样品进行物理或化学裂解,从中提取出RNA和RNA结合蛋白。如果需要检测核糖体RNA(rRNA)的结合情况,则需要进行去除rRNA的处理。
2、免疫共沉淀
将提取出的RNA和RNA结合蛋白与特异性的抗体结合。为了充分保证实验结果的可靠性,需要选择合适的抗体进行共沉淀,而且要仔细控制反应条件。对于没有可靠抗体的蛋白,可以使用His标签等融合标记来进行共沉淀。之后加入蛋白A/G磁珠进行免疫亲和。
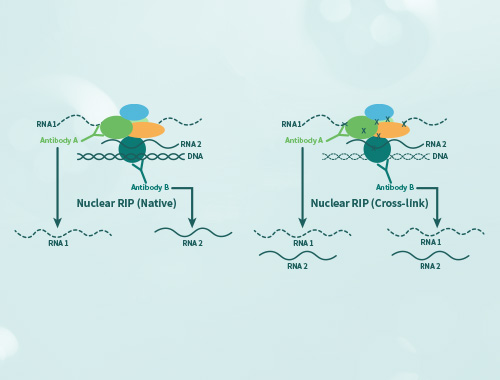
3、洗涤
使用不同的缓冲液进行多次洗涤,以去除非特异性结合的其他组分。通常使用高盐环境来除去非特异性蛋白质,使抗体与特异性结合的蛋白质保持稳定。
洗涤后还要检查磁珠是否干净,以免影响后续的实验步骤。
4、RNA结合蛋白和靶RNA的分离
将沉淀的RNA结合蛋白与靶RNA进行分离。分离可以通过化学或物理方法完成,例如使用三氯乙酸(TCA)沉淀或使用高渗盐的酸性酚/氯仿提取法。
5 、RNA逆转录
使用配对的逆转录酶和引物,将RNA逆转录成cDNA,并且进行PCR扩增。可以使用DNase等酶对RNA进行降解处理,同时还可在cDNA合成过程中加入冗余引物来去除被逆转录的RNA模板。
6、qPCR分析
将得到的cDNA进行实时荧光定量PCR(qPCR)分析,以确定RNA结合蛋白与靶RNA之间的相互作用情况。在实行qPCR实验前,最好先进行实验优化,包括最佳引物浓度、最佳反应体系等,以保证最终的结果准确可靠。
总结:RIP-qPCR是一种用于研究RNA结合蛋白与靶RNA相互作用的实验方法,其步骤主要包括细胞裂解、免疫共沉淀、洗涤、RNA结合蛋白和靶RNA的分离、RNA逆转录和qPCR分析。实验过程中需要仔细控制反应条件,以保证实验结果准确可靠。










