表观遗传 | 超级增强子相关lncRNA如何影响肝癌
前言:增强子(enhancer),是距离启动子较远距离的一段可与蛋白质(反式作用因子,trans-acting factor)结合的区域,可以被转录因子等蛋白结合从而激活基因转录。增强子在不同类型细胞以及不同发育阶段会有不同活性,并且与转录因子(Transcript Factor, TF)等蛋白的结合也具有特异性,因此增强子是调控基因特异性表达重要的顺式作用元件(Cis-regulatory element)(Fig 1)[1]。
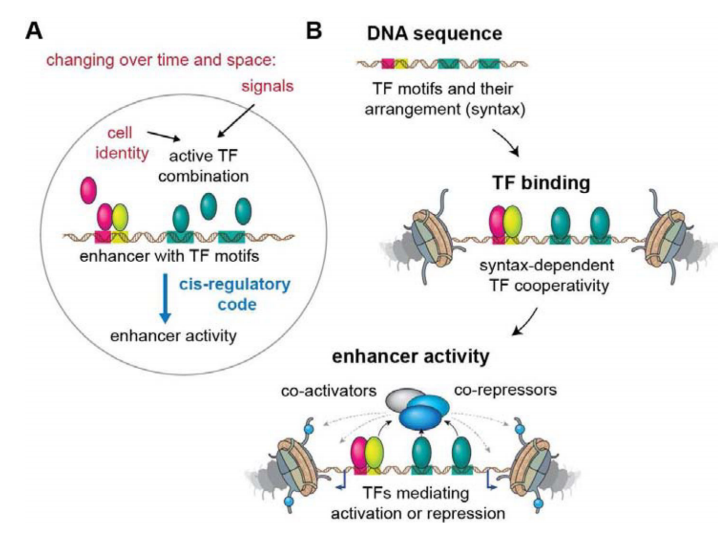
Figure 1 转录因子调控增强子活性示意图[1]。(A)在不同时空中,特定TF通过转录或细胞外信号进行识别motif序列与增强子结合并激活增强子。(B)顺式调控元件含有特定的TF基序(motif)。TF招募共激活因子或共阻遏因子,其调控增强子的活性。
在后生动物中,启动子具有H3K4me3标记,同样增强子在基因组上也有特殊的身份标记——增强子特征是具有染色质开放性和组蛋白H3K4me1修饰,活性增强子(Active Enhancer)具有H3K27ac标记,静态/起始增强子(Poised/Primed Enhancer)缺少H3K27ac标记(Fig 2)[2]。因此我们可以根据CUT&TAG、ChIP-seq和ATAC-seq等技术鉴定增强子位点。

Figure 2 顺式调控元件基因组特征[2]。增强子特征是具有染色质开放性和组蛋白H3K4me1修饰,活性增强子(Active Enhancer)具有H3K27ac标记,静态/起始增强子(Poised/Primed Enhancer)缺少H3K27ac标记。
随着对增强子在转录调控作用研究的深入,Richard A. Young团队于2013年提出超级增强子概念——超级增强子(Super-enhancer, SE)比普通增强子(Typical enhancer,TE)具有更高的转录激活相关组蛋白修饰(H3K27ac、H3Kme1等)、Mediator复合体和BRD4蛋白(Bromodomain containing 4)及转录因子的富集密度,超级增强子区域长度通常可达8-20 Kb,远高于普通增强子的200-300 bp长度[3]。长非编码RNA(lncRNA)的异常表达已在多种癌症中屡见报道,而SE与lncRNA关联的新研究思路近年来初露锋芒。SE-lncRNA就是超级增强子关联的lncRNA,它紧靠SE并且其转录活性受到SE的调控。这里小编分享2篇近期的肝癌方面SE-lncRNA高水平研究,帮助各位老师了解SE-lncRNA课题研究思路。
一、超级增强子驱动的lncRNA-Daw通过激活Wnt/b-catenin途径促进肝癌细胞增殖
广州中医药大学张锦芳团队2021年在Mol Ther Nucleic Acids. 发表Super-enhancer-driven lncRNA-DAW promotes liver cancer cell proliferation through activation of Wnt/β-catenin pathway一文揭示了lncRNA-DAW在肝癌发展中的作用机制[4]。
1、首先比较各人体组织中H3K27ac ChIP-seq数据,以筛选肝部特异性SE(Fig 3)。与其他实体瘤相似,在肝细胞癌(HCC)中也经常检测到基因组DNA拷贝数的扩增,包括20q13.12的扩增,既往研究表明,20q13.12片段的获得与肝癌患者的肝转移、总生存期及不良预后显著相关。而20q13.12片段在HCC中有比正常细胞更高的H3K4me1和H3K27ac信号,该位点可能含有肝脏特异性超级增强子。

Figure 3 人各组织H3K27ac数据[4]。(A) 一组正常人体组织中lncRNA DAW(LINC01430)基因座的H3K27ac-ChIP-seq数据。黄色区域表示lncRNA DAW的位点。(B) 肝细胞中lncRNA DAW基因座的超级增强子组蛋白标记的ChIP-seq数据。
2、在该SE区域鉴定到8个此前未报道的lncRNA,根据RNA-seq数据,此前被称为lncRNA-DAW的lncRNA在肝组织中高表达。比较肝细胞癌(HCC)和正常细胞中表达量,发现lncRNA-DAW显著上调。因此,作者将它确定后HCC候选靶点lncRNA进行进一步细胞和动物实验。
3、超级增强子经常被各种转录因子占据以发挥作用,作者筛选lncRNA-DAW所在SE依据UCSC基因组浏览器和Cistrome数据库候选结合的TF,发现该基因位点与肝细胞核因子家族成员HNF4G结合。根据ChIP-seq实验、双荧光素酶实验和RT-PCR实验,HNF4G 与候选区域中的SE-1 和 SE2结合,并且HNF4G促进lncRNA-DAW表达。
4、作者通过体外过表达和裸鼠实验证明lncRNA-DAW促进癌细胞增殖和转移。对lncRNA-DAW过表达细胞进行RNA-seq,对癌症相关通路中选择候选基因进行RT-qPCR实验和siRNA干扰实验,Wnt2基因表现受到lncRNA-DAW调控。
5、根据此前报道lncRNA可能与多梳蛋白抑制复合物(Polycomb repressive complexes, PRC)结合,作者发现PRC核心成分EZH2会抑制Wnt2表达。根据ChIP-seq实验发现,EZH2会结合Wnt2启动子从而抑制转录。
6、作者通过RIP-qPCR验证EZH2会结合lncRNA-DAW,lncRNA-DAW过表达后EZH2对Wnt2启动子抑制效果下降。由于过去报道多种lncRNA可以调节RNA结合蛋白的磷酸化和稳定性,于是作者通过WB验证了lncRNA-DAW的瞬时过表达以剂量依赖性方式降低内源性EZH2蛋白量。
7、依据细胞和动物实验作者证明Wnt2激活了Wnt/β-catenin通路促进癌细胞增殖。作者根据50对肝癌表达谱数据发现HCC标本中lncRNA-DAW和Wnt2的表达之间的显著正相关,lncRNA-DAW和Wnt2表达的增加可能是人类HCC组织中的常见事件,表明lncRNA-DAW和Wnt2可能作为肝癌患者的假定治疗靶点(Fig 4)。
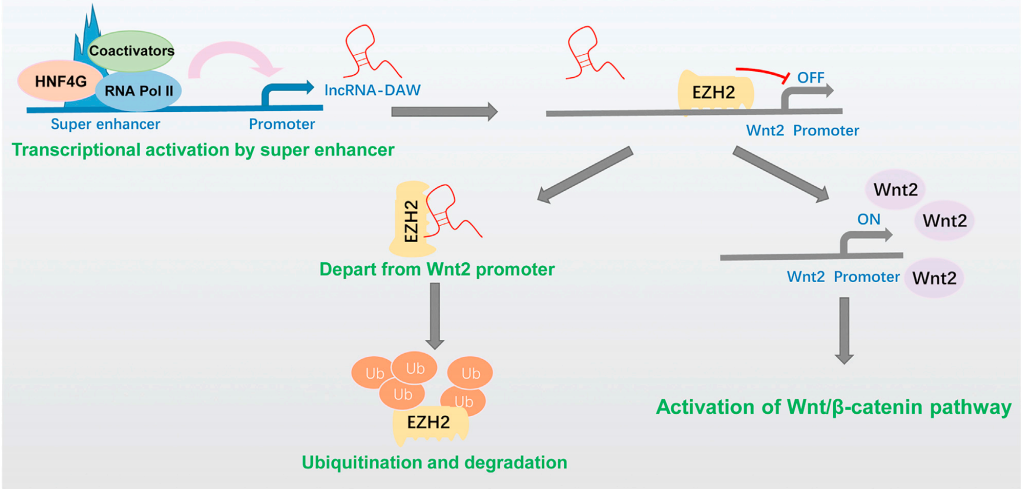
Figure 4 lncRNA-DAW调控Wnt2促进肝癌细胞增殖示意图[4]。
二、lncRNA FASRL促进脂肪酸生物合成并加剧肝癌进展
中山大学彭丽团队于去年在Advanced Science上发表了题为“Upregulation of Superenhancer-Driven LncRNA FASRL by USF1 Promotes De Novo Fatty Acid Biosynthesis to Exacerbate Hepatocellular Carcinoma”文章。研究发现了一种名为FASRL 的lncRNA表达受超级增强子调控,表达上调后可以促进脂肪酸的从头合成,从而促进肝细胞癌的发生[5]。
1、作者通过HepG2、Huh7、PLC、LM3(四种肝癌细胞系)和正常肝组织的H3K27ac数据,筛选并鉴定了由HCC细胞中异常超级增强子调节的lncRNA(Fig 5)。作者鉴定到31个肝癌特异性SE相关的lncRNA,其中3个lncRNA在HCC样本中上调,其高表达与HCC患者预后较差相关。作者将其中表达差异最大的1个lncRNA命名为 “FASRL”并作为候选肝癌靶点SE-lncRNA。FASRL位于16号染色体上,长度为15551 bp,包含两个外显子和一个内含子。
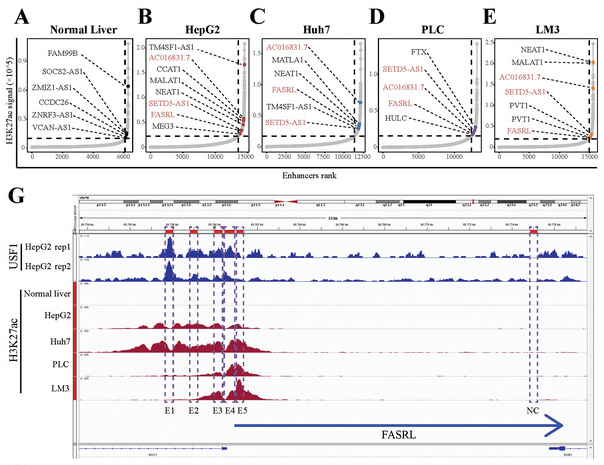
Figure 5 筛选肝癌特异性超级增强子[5]。A-E. 正常肝组织和四种肝癌细胞系超级增强子,FASRL是四种癌细胞系共有的特异性超级增强子,虚线代表ROSE软件判断SE的阈值。G. FASRL处H3K27ac信号,虚线为增强子位点,四种肝癌细胞中存在较高H3K27ac信号,而正常肝细胞中不存在。
2、作者将FASRL的五个增强子序列进行双荧光素酶实验,增强子E4对FASRL表达增强效果最强。为了探究SE调控FASRL表达上游机制,作者对FASRL序列进行转录因子motif分析,把将排名前十的TF表达量与FASRL表达量进行相关性分析,相关性最高的是热休克转录因子1(HSF1)。之后通过siRNA和ChIP-qPCR实验证明TF USF1可以通过与HCC中的SE结合来转录驱动FASRL表达(Fig 6)。
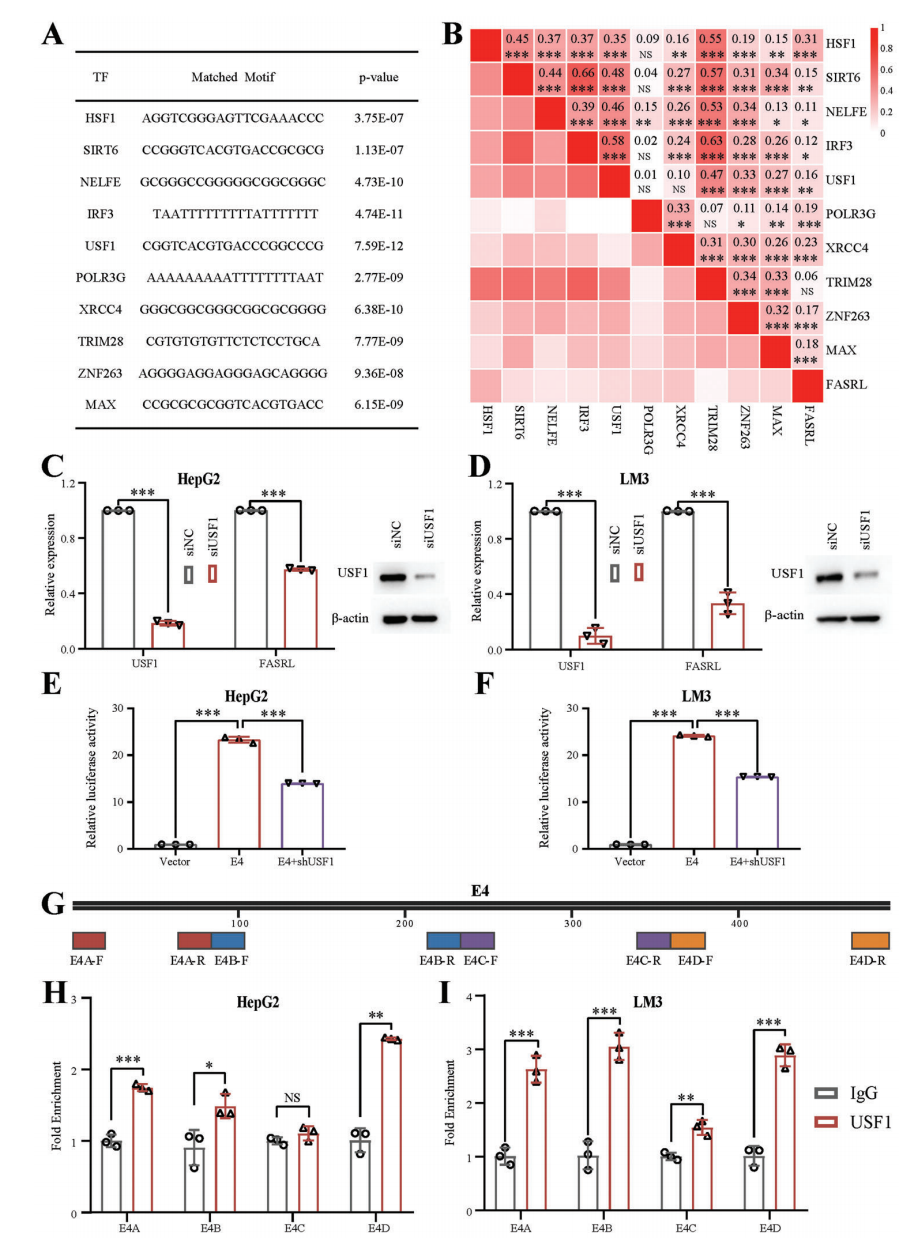
Figure 6 验证HSF1是调控FASRL的转录因子[5]。A. FASRL区域序列前10可能结合的TF motif序列。B. 10个TF和FASRL表达相关性。C,D. siRNA干扰USF1后HepG2和LM3细胞系中USF1和FASRL表达量和蛋白含量。E,F. 双荧光素酶实验显示了shRNA敲低USF1后E4的转录活性下降。G. 在E4序列上设计了4对ChIP-qPCR引物,H图为USF1结合SE的ChIP-qPCR结果。
3、作者进行体外实验验证FASRL是否能够影响肝癌细胞进展。通过siRNA敲低FASRL后细胞增殖、细胞迁移、细胞凋亡证明FASRL可以促进HCC细胞的致癌生长。之后将稳定转染shFASRL或shNC的HCC细胞系皮下注射到裸鼠中,观测到shFASRL显著减缓了裸鼠中肿瘤细胞的增殖。
4、为了揭示FASRL的特定下游调控途径,作者依据siFASRL转录组数据进行基因集富集分析(GSEA),FASRL敲低与脂肪酸(FA)途径相关基因的表达呈显著负相关,之后进行RT-qPCR和WB实验验证了这个发现(Fig 7)。
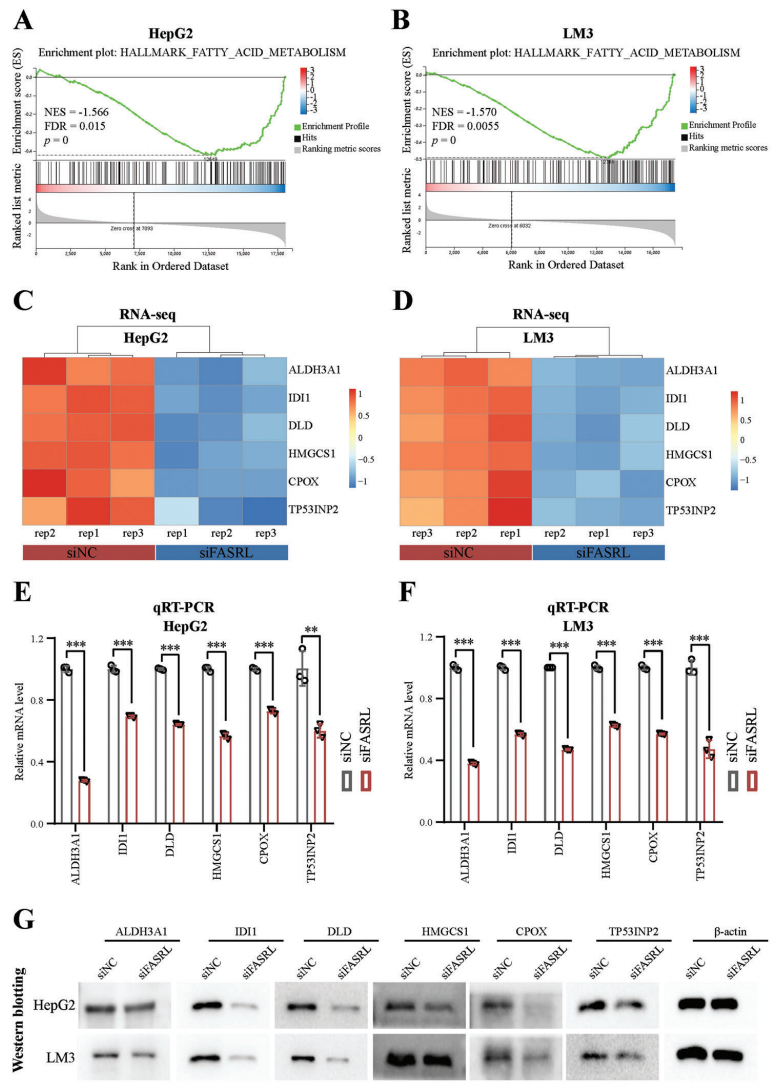
Figure 7 FASRL参与HCC脂肪酸代谢通路[5]。A,B 敲除FASRL表达后表达改变的基因在脂肪酸(FA)代谢通路中富集。C,D qRT-PCR显示FASRL表达下调后,FA代谢通路中表达显著改变的基因表达量热图。E,F FASRL的表达下调后,FA代谢通路中基因的mRNA表达显著下调。G. WB显示FASRL 表达在 HepG2 和 LM3 细胞系中敲低后 FA 代谢途径中基因的蛋白质含量改变。
5、为了进一步探索与FASRL直接相互作用的蛋白质,作者使用RNA Pull Down-MS鉴定与FASRL直接相互作用的RNA结合蛋白,MS结果显示乙酰辅酶A羧化酶1(ACACA)直接与FASRL结合,之后进行RIP-qPCR、FISH、IHC和WB进行进一步验证。先前的研究表明,ACACA (Ser79) 的磷酸化抑制其酶活性并减少 FA 合成,而本研究发现敲低FASRL表达增加了磷酸化ACACA的水平,FASRL与ACACA结合并抑制其磷酸化,从而增加了FA的合成。
6、ACACA是脂质代谢中的重要酶,是控制从头FA生物合成的中心酶。作者用同位素13C6-标记葡萄糖追踪FA合成,证明敲低FASRL抑制了从头FA合成,另外FASRL敲低显著降低了甘油三酯(TG)含量。之后通过油红O染色实验和对脂滴荧光标记进一步验证了在敲低FASRL表达后HCC细胞中的脂滴数量显著减少。
7、综上所述,这项工作发现USF1可以通过SE驱动FASRL表达,FASRL和ACACA之间的相互作用增加FA合成,以促进肿瘤进展(Fig 8)。作者期望未来开发抑制TF USF1或SE的药物来降低FASRL的表达或抑制FASRL与ACACA的结合以减少FA合成,最终达到抑制HCC的目的。
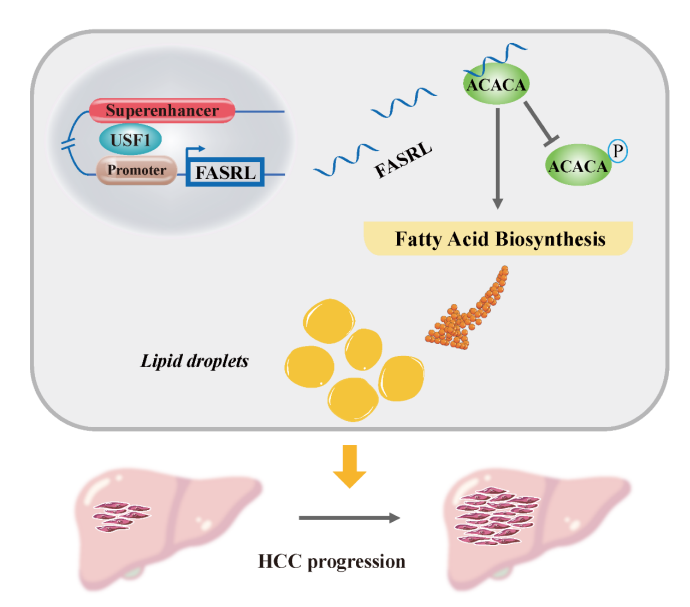
Figure 8 SE-lncRNA FASRL影响肝癌进展机制[5]。
总结以上两篇文章中涉及实验有ChIP-seq、ChIP-qPCR、RNA-seq、RIP-qPCR、RNA Pull down-MS、双荧光素酶报告基因、CoIP-MS、细胞功能实验、动物建模实验等。对于这些实验,武汉金开瑞生物均有大量项目经验,可助力您课题研究,欢迎各位老师咨询!

参考文献:
[1] Zeitlinger J. Seven myths of how transcription factors read the cis-regulatory code. Curr Opin Syst Biol. 2020;23:22-31. doi:10.1016/j.coisb.2020.08.002
[2] Preissl S, Gaulton KJ, Ren B. Characterizing cis-regulatory elements using single-cell epigenomics. Nat Rev Genet. 2023;24(1):21-43. doi:10.1038/s41576-022-00509-1
[3] Whyte WA, Orlando DA, Hnisz D, et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 2013;153(2):307-319. doi:10.1016/j.cell.2013.03.035
[4] Liang W, Shi C, Hong W, et al. Super-enhancer-driven lncRNA-DAW promotes liver cancer cell proliferation through activation of Wnt/β-catenin pathway. Mol Ther Nucleic Acids. 2021;26:1351-1363. Published 2021 Nov 3. doi:10.1016/j.omtn.2021.10.028
[5] Peng JY, Cai DK, Zeng RL, et al. Upregulation of Superenhancer-Driven LncRNA FASRL by USF1 Promotes De Novo Fatty Acid Biosynthesis to Exacerbate Hepatocellular Carcinoma [published online ahead of print, 2022 Oct 28]. Adv Sci (Weinh). 2022;10(1):e2204711. doi:10.1002/advs.202204711
最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










