文献阅读:一分钟教你看懂火山图
一张火山图,其实就是一张“散点图”,它是生物信息学中用于高通量数据差异分析(如转录组学、蛋白质组学)的核心可视化工具。通过整合统计学显著性与生物学变化幅度,将成千上万的分子(如基因、蛋白)直观地呈现在一张散点图上,从而帮助研究者快速、高效地识别出在两组样本间发生显著变化的关键目标。
理解火山图的口诀:横着看差异,竖着看(统计学)意义
1.横轴(Log₂ Fold Change):表示分子表达量的差异倍数。零点右侧代表上调,左侧代表下调;偏离零点越远,说明基因的表达变化越大。
2.纵轴(-Log₁₀ P-value):表示差异的统计学显著性。这个轴代表了这种变化的可靠性,通常用P值来表示。 P值越小,说明这种变化不是偶然发生的可能性越大,也就是越可信。因为P值通常很小,所以为了方便在图表上表示,一般会对P值取负对数(比如-log10(P值))。这样,纵轴上的点越高,说明这种变化越显著。
3.阈值线:图上通常通过两条阈值线(垂直的倍数变化阈值与水平的显著性阈值)划分出三个关键区域:显著上调区、显著下调区以及非显著区。
左侧显著区域(标蓝): 显著下调的点。Log2FC < -阈值 且 -Log10(P-value) > 阈值。
右侧显著区域(标绿): 显著上调的点。Log2FC > 阈值 且 -Log10(P-value) > 阈值。
中间灰色不显著区域:变化不大或统计不显著的点,是图中的“背景”。
只有同时跨越两条阈值的点(图中蓝色和绿色高亮的点),才被认为是具有生物学意义的差异表达分子。
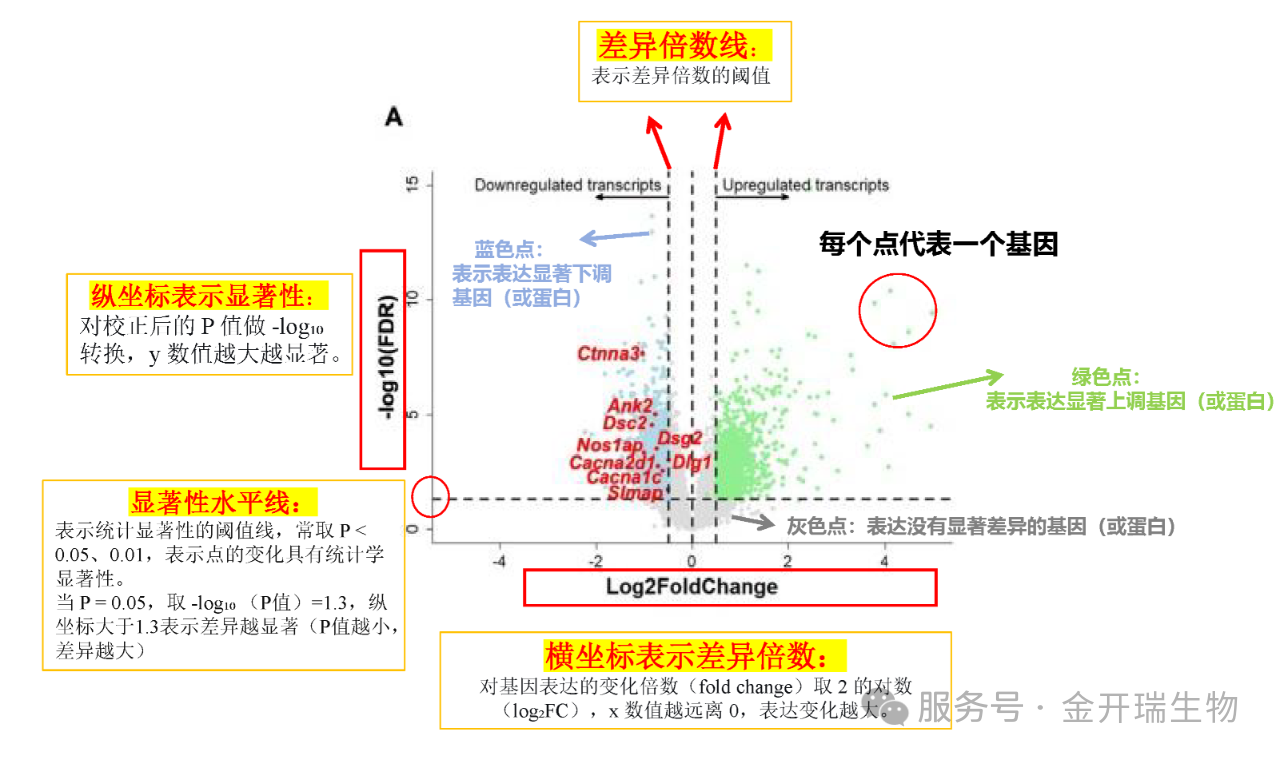
图1.火山图解析
去年6月份发表在顶刊《science》杂志上的一篇题为“Defining the KRAS- and ERK-dependent transcriptome in KRAS-mutant cancers”的文章中展示一张比较特殊的火山图:
跟常规的火山图相比,这个图在底部展示了两个特征基因在基因集中的分布情况,竖线表示基因集中的基因在横轴即所有基因按照 FC 值排序的基因中的位置,就是GSEA结果中的那种排序竖线。而且在这个火山图中,通过颜色填充区分特征基因和非特征基因,使得关键基因集中分布的区域一目了然。
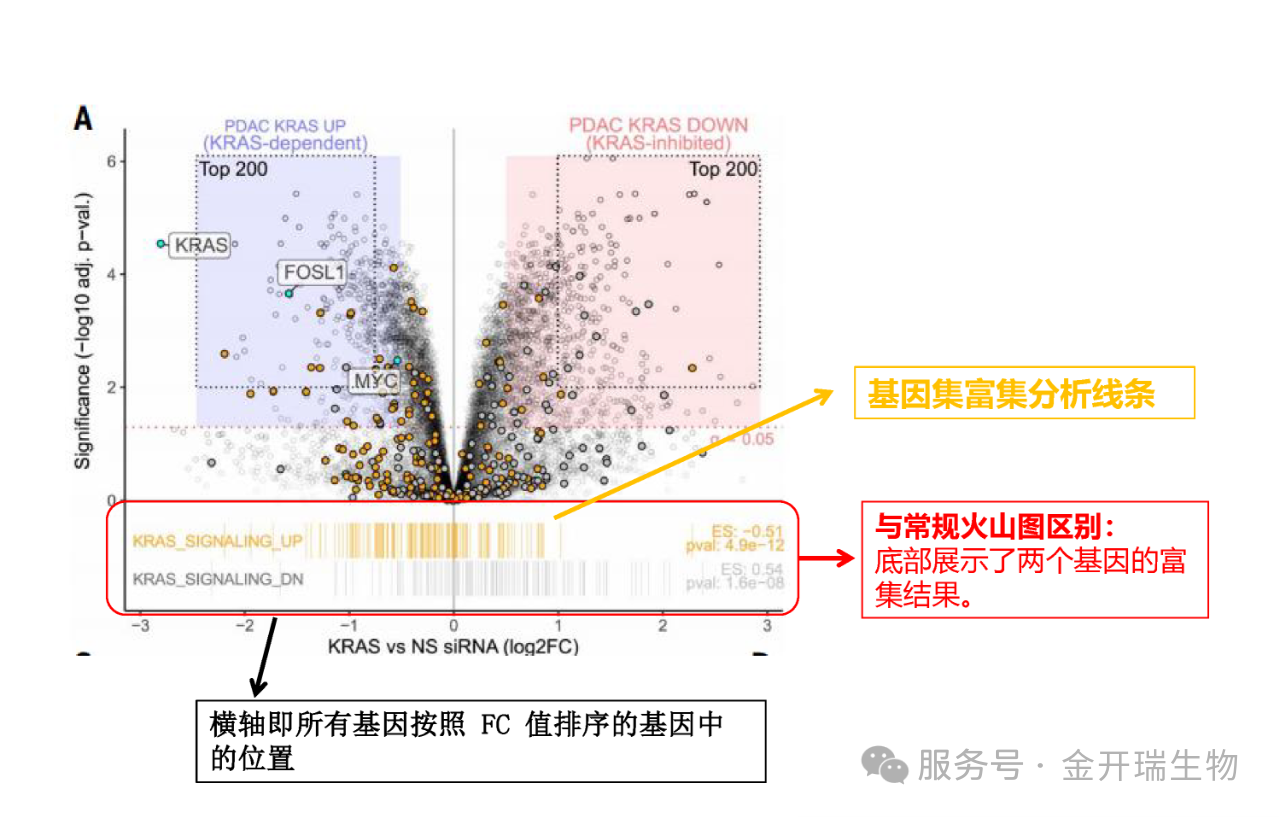
图2.差异基因表达分析显示所有十个簇中基因均存在上调和下调。调整后的p值<0.01以红色表示,调整后p值≥0.01以黑色表示。
还有一篇发表在《CELL》杂志上,文献标题为“A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart”的文章,为了识别差异表达的基因,使用Seurat软件包中的FindAllMarkers函数(设置:min.pct = 0.25,thresh.use = 0.25)对各个cluster与其他所有cluster进行了成对比较。这种采用多比较组差异火山图( 仅保留log2FC)是一种目前比较新颖的火山图绘制方式。
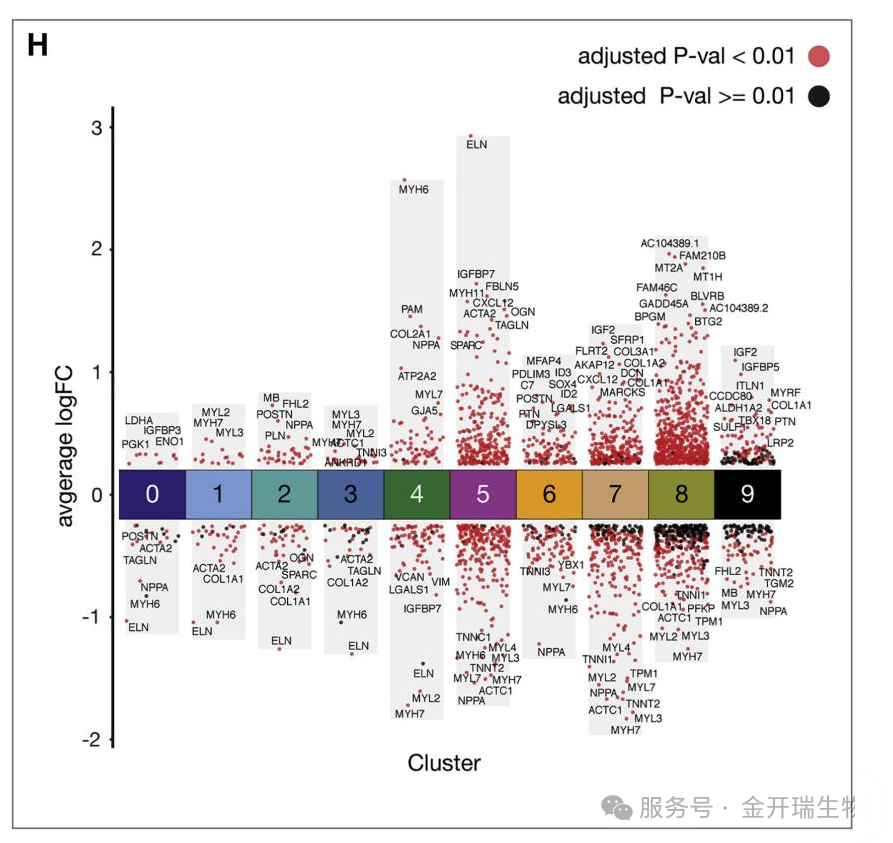
图3.差异基因表达分析展示所有十个细胞簇中的上调与下调基因。校正p值<0.01的基因以红色标注,而校正p值≥0.01的基因以黑色标注。
总而言之,火山图作为差异表达分析的“地图”,通过整合差异倍数与统计显著性,帮助研究者高效锁定关键分子。随着组学研究的深入与可视化技术的发展,火山图在高水平论文中也展现出丰富多元的表现形式。例如,除了经典的双阈值散点图外,还出现了整合GSEA排序结果的增强火山图(如《Science》文章中所示),在底部标记特征基因集的分布位置;以及多比较组火山图(如《Cell》文章中采用),在同一坐标中同时展示多个条件下的表达变化。这些创新不仅提升了信息密度,也增强了结果的解释力。
因此,掌握火山图的基本读图要诀——“横看差异,纵看意义”——是理解文献与开展分析的基础,而在此基础上,关注并尝试探索适合自身研究问题的新型可视化形式,将有助于更清晰、更高效地传递科学发现。
参考文献:
[1]Marina Cerrone,et al. Role of plakophilin-2 expression on exercise-related progression of arrhythmogenic right ventricular cardiomyopathy: a translational study, European Heart Journal, Volume 43, Issue 12, 21 March 2022, Pages 1251–1264.
[2]Hibshman PS, et al. Defining the MYC-regulated transcriptome and kinome that support KRAS- and ERK-dependent growth of pancreatic cancer. Sci Signal. 2025 Sep 30;18(906):eadu7145.
[3]Asp M,et al. A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart. Cell. 2019 Dec 12;179(7):1647-1660.e19.
最新动态
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图
-
12.10
RNA-蛋白互作研究全景解析:从circPOLR2A案例看机制探索之路
-
12.10
神奇的“植物外泌体”?铁皮石斛衍生的纳米囊泡登顶顶刊,揭示皮肤再生新密码!
-
11.20
Journal of Pharmaceutical Analysis | 生姜外泌体作为姜黄素递送系统在治疗溃疡性结肠炎中的应用
-
11.20
J Nanobiotechnology | 叶酸修饰的生姜衍生的细胞外囊泡可以重塑免疫微环境,靶向治疗类风湿性关节炎
-
11.20
WB实验翻车无数次?可能从一开始,你的抗体就选错了!
-
11.20
从“核心”到“网络”:如何以已知基因为锚点,绘制其上下游通路图谱!










