技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
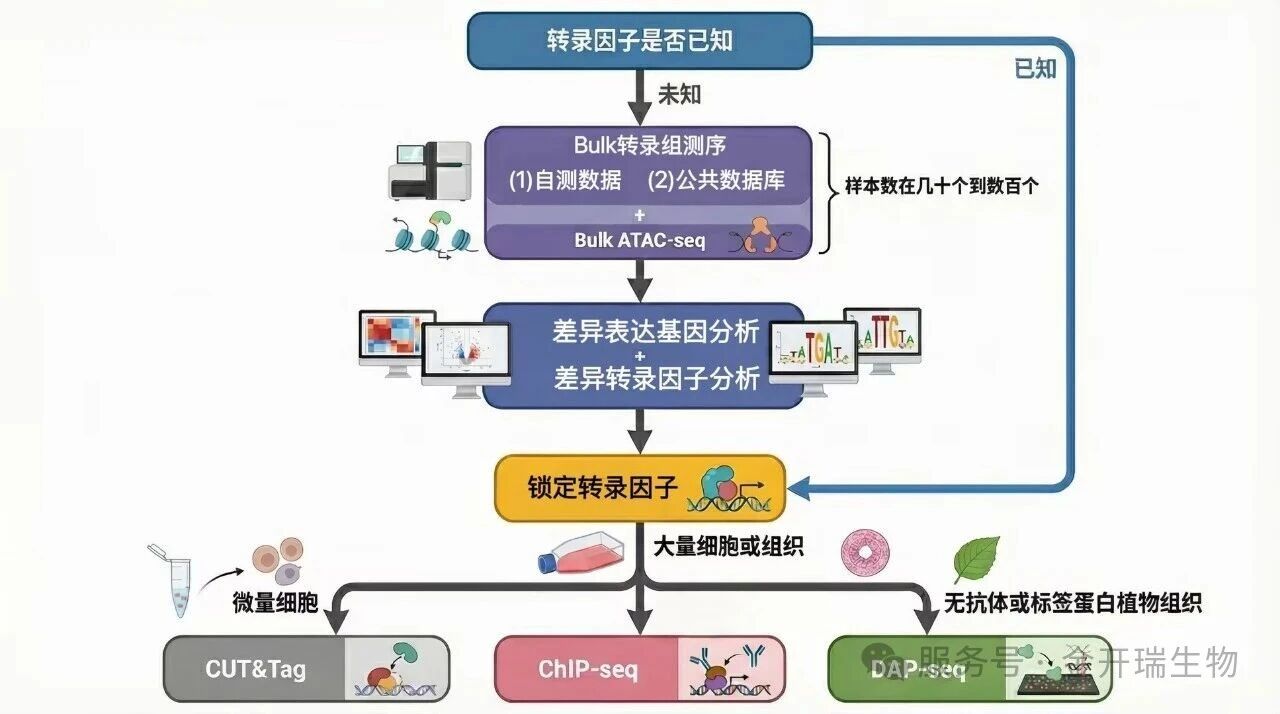
近年来,表观遗传学研究技术的快速发展为解析染色质动态调控机制提供了多维工具。其中,CUT&Tag、ATAC-seq、ChIP-seq和DAP-seq四种技术因其独特的技术优势被广泛应用于染色质可及性、蛋白质-DNA互作及表观修饰的研究中。通过技术联用,研究者能够从不同维度揭示染色质调控的复杂机制,尤其在解析细胞异质性、发育动态和疾病机制方面展现出强大的协同效应。
1、技术联用的创新思路
CUT&Tag作为ChIP-seq的高灵敏度替代方案,通过抗体引导的Tn5靶向切割实现了低细胞起始量(低至20,000细胞)和高信噪比的表观标记检测。其与ATAC-seq的联用可同时捕获染色质开放状态(ATAC-seq)和特定组蛋白修饰(如H3K27ac或H3K27me3)的空间共定位,例如在少突胶质细胞谱系研究中,nano-CUT&Tag技术通过整合染色质可及性(ATAC)、活性增强子标记(H3K27ac)和抑制性标记(H3K27me3)的三模态数据,揭示了H3K27me3在分化过程中对基因模块的时序性抑制规律。这种多组学联用策略不仅提升了细胞分型分辨率,还通过“染色质速度”分析预测了表观状态的动态转换。
ChIP-seq与ATAC-seq的经典组合则常用于验证转录因子结合位点与染色质开放区域的关联性。例如,在ENCODE项目中,H3K27ac的ChIP-seq峰值与ATAC-seq信号高度重叠,共同标记了活跃的增强子和启动子区域。而DAP-seq(DNA亲和纯化测序)作为体外高通量筛选技术,可快速鉴定转录因子的DNA结合motif,其数据常被用于指导ChIP-seq或CUT&Tag的靶向分析,尤其在植物研究中显著提高了转录因子调控网络的预测效率。
2、高影响力文献中的技术联用解析
文献1:近期,浙江大学医学院附属第一医院黄河教授与钱鹏旭教授团队在影响因子40+的著名期刊《Journal of Hematology & Oncology》上发表重要研究,题为“BRD4 acts as a transcriptional repressor of RhoB to inhibit terminal erythropoiesis”。该研究主要通过联合RNA-seq、ATAC-seq、CUT&Tag、ChIP-qPCR、Co-IP等实验手段,首次揭示BRD4在终末红细胞生成中发挥“刹车”作用。研究进一步阐明BRD4通过非经典转录抑制机制(不依赖CDK9),协同组蛋白甲基转移酶EHMT1/2沉默小G蛋白RhoB的表达,从而阻碍红细胞成熟进程。这一发现为深入解析红细胞生成的表观遗传调控网络及相关疾病的治疗策略提供了新的理论视角。
|
实验技术 |
应用思路与目的 |
|
高通量小分子筛选 |
从348种靶向表观与转录调控的化合物中,快速筛选出能促进红细胞终末分化的候选靶点。 |
|
RNA-seq |
分析BRD4抑制后全转录组变化,识别差异表达基因。 |
|
ATAC-seq |
检测染色质开放程度,评估BRD4抑制对染色质结构的全局影响。 |
|
CUT&Tag |
高分辨率绘制BRD4在全基因组的结合图谱,并联合RNA-seq识别其直接调控的靶基因。 |
|
ChIP-qPCR |
验证BRD4在 RhoB 基因启动子区的直接结合,并分析Pol2磷酸化状态。 |
|
Co-IP |
检测BRD4与潜在共抑制因子的蛋白质相互作用。 |
|
流式细胞术 |
定量红细胞表面标志物(CD36、GPA、CD45等)的变化,评估分化阶段与效率。 |
研究人员通过RNA-seq与ATAC-seq联合分析发现,抑制BRD4后,红细胞成熟相关基因(如HDAC5、FOXO1)表达上调,而增殖相关基因(如MYC、CDK6)表达下调,同时染色质可及性降低,提示BRD4抑制促进了染色质凝缩和红细胞成熟进程(图A–F)。为验证BRD4是否直接调控RhoB,研究人员进行了ChIP-qPCR实验,证实BRD4直接结合在RHOB基因座附近,且该结合在BRD4抑制后显著减少,而CDK9在该区域无结合,说明BRD4对RhoB的抑制不依赖于其经典搭档CDK9(图I–K)。
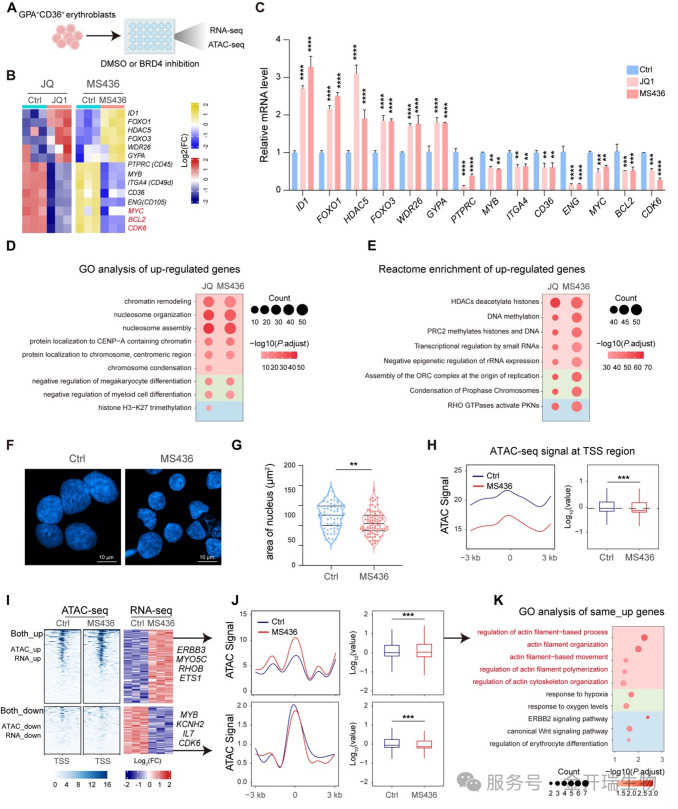
RNA-seq和ATAC-seq的联合分析表明,BRD4抑制加速终末红细胞生成
进一步通过CUT&Tag技术直接检测BRD4在全基因组的结合情况,发现BRD4抑制后其结合减少的区域与表达上升的基因高度重叠,其中小G蛋白RhoB的表达显著上调,提示BRO4可能通过抑制RhoB发挥功能(图A–F)。为验证BRD4是否直接调控RhoB,研究人员进行了ChIP-qPCR实验,证实BRD4直接结合在RHOB基因座附近,且该结合在BRD4抑制后显著减少,而CDK9在该区域无结合,说明BRD4对RhoB的抑制不依赖于其经典搭档CDK9(图I–K)。最后通过Co-IP实验发现,BRD4与组蛋白甲基转移酶EHMT1/2存在相互作用,提示BRD4可能通过招募EHMT1/2催化抑制性组蛋白标记H3K9me2,从而抑制RhoB表达。
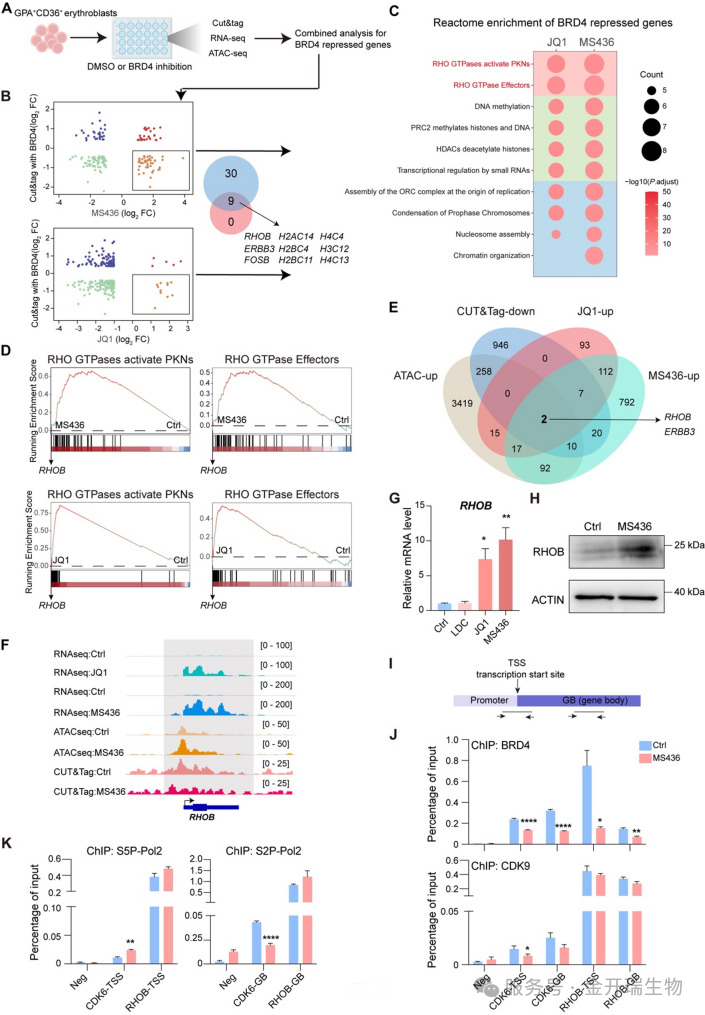
BRD4抑制RhoB的表达:RNA-seq、ATAC-seq和CUT&Tag三种技术联合分析
文献2:中国医学科学院北京协和医学院基础医学研究所和北京脑科学与类脑研究所联合在《Nature》期刊上发表了一篇题为“Oncogenic fusions converge on shared mechanisms in initiating astroblastoma”的文章,系统揭示了致癌融合蛋白MN1-BEND2驱动星形母细胞瘤(ABM)的分子机制。该研究综合利用ChIP-seq、ATAC-seq等表观多组学技术,首次系统阐明:ABM相关融合基因通过识别共同DNA基序→重塑染色质可及性→激活PDGFRα通路这一统一机制驱动肿瘤发生,并明确腹侧端脑神经前体细胞为肿瘤起源,OLIG2是介导细胞凋亡逃逸的关键因子。
研究人员首先通过 scRNA-seq 在单细胞精度上观察最终的转录输出与细胞表型。分析发现,表达MN1-BEND2的腹侧端脑神经祖细胞,其转录状态发生根本性重塑:神经干细胞样群体扩增、分化受阻,并异常高表达PDGFRA等ABM特征基因。
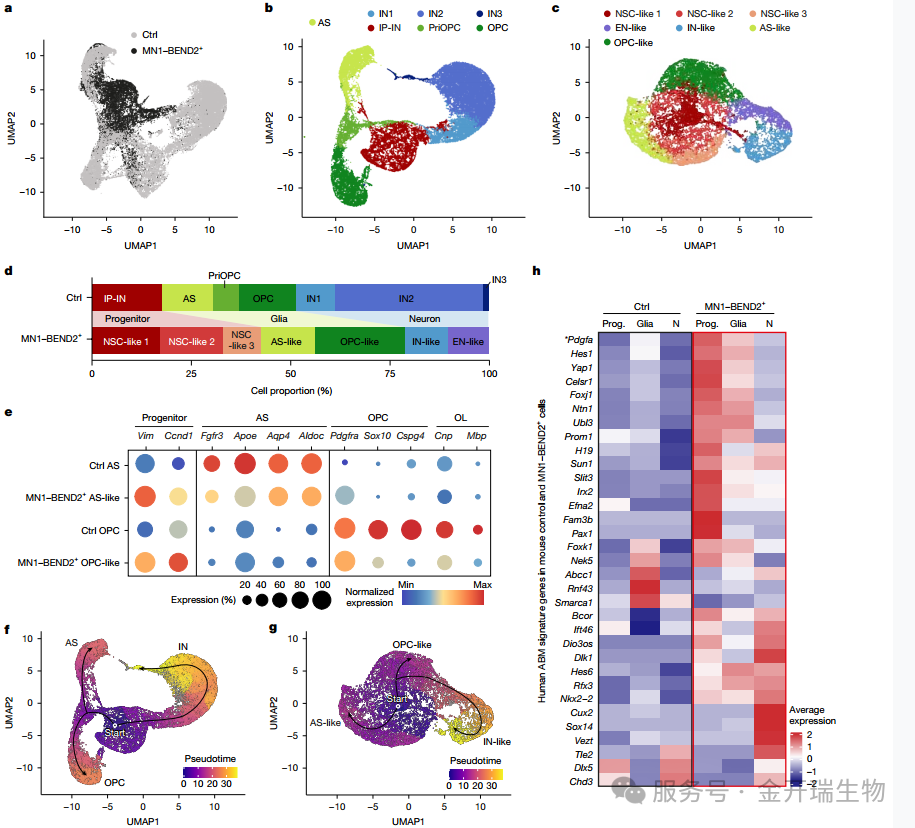
MN1-BEND2损害腹侧端脑祖细胞的发育并诱导ABM相关的转录特征
接着运用 CUT&Tag 技术,在表达MN1-BEND2的小鼠神经祖细胞中,高灵敏度地绘制了该融合蛋白的DNA结合图谱。结果发现,MN1-BEND2特异性结合在包含CGTT基序的基因组区域,并直接靶向 Pdgfra、Olig2 和 Pdgfa 等关键原癌基因的调控元件。为验证融合蛋白的结合是否真正影响了染色质状态,研究者对同一群细胞进行了 ATAC-seq 分析。结果显示,在MN1-BEND2结合的区域,染色质可及性显著增加。这表明融合蛋白不仅结合DNA,更直接打开了靶基因的染色质,使其处于易被激活的状态。这一步与CUT&Tag结果相互印证。
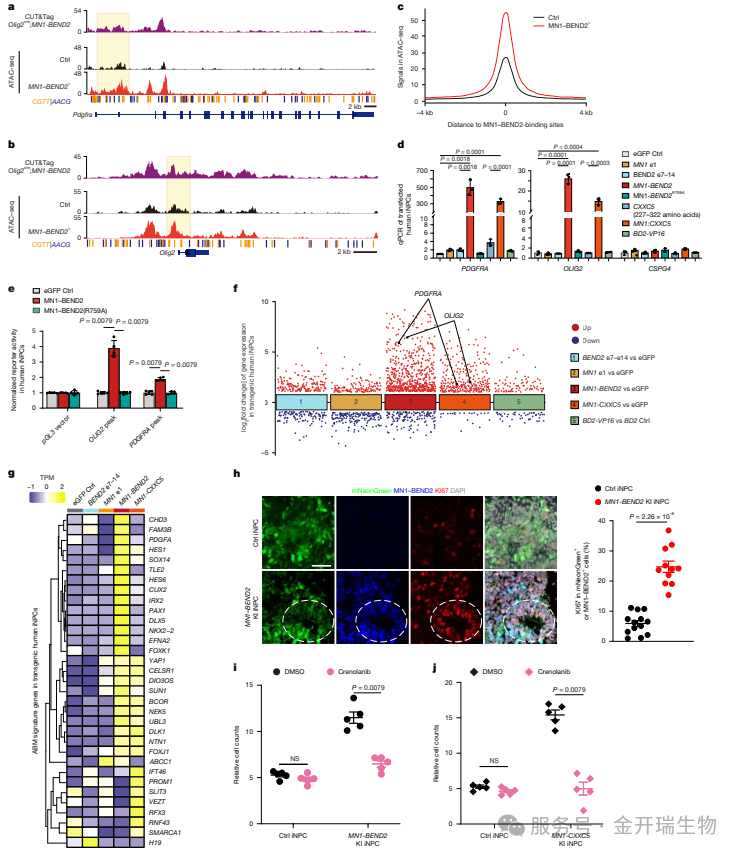
融合蛋白直接激活PDGFRA引发恶性肿瘤
3、技术联用的验证逻辑
在上述研究中,多技术联用的核心价值在于交叉验证与数据互补。例如,CUT&Tag的高灵敏度可检测低频细胞群体的表观特征(如肿瘤微环境中的稀有亚群),而ATAC-seq的全局染色质图谱则为这些标记提供了开放状态的上下文。在数据分析层面,研究者常采用以下策略:
(1) 通过peak重叠分析(如Bedtools)确认技术间的一致性;
(2) 利用差异峰与差异可及性区域的共现性推断功能关联;
(3) 结合motif富集(如HOMER)从DAP-seq数据中预测转录因子结合位点,再通过CUT&Tag/ChIP-seq验证其体内结合活性。
这种多维验证体系显著提高了结论的可靠性,尤其在解析复杂调控网络时表现出不可替代的优势。
未来,随着单细胞多组学技术的发展,CUT&Tag与ATAC-seq的整合将更趋标准化。例如,空间表观组学技术(spatial-CUT&Tag)已实现组织原位染色质修饰的微米级分辨率成像,为发育和疾病研究提供了空间维度的新见解。而纳米孔测序与CUT&Tag的结合(scNanoSeq-CUT&Tag)则进一步突破了重复序列和复杂基因组区域的检测瓶颈。这些创新联用策略将持续推动表观遗传学从描述性研究向机制解析和临床转化的跨越
表观组学的多维视角与联合应用,正为我们揭示更精细的基因调控网络。无论是ChIP-seq、ATAC-seq,还是CUT&Tag与DAP-seq的灵活搭配,金开瑞以专业的技术平台、深度的生信分析及全程课题支持,助您打通数据到生物学意义的最后一公里。
最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










