科研干货 | 从什么渠道找ChIP抗体更迅速、准确、可靠呢?
染色体免疫共沉淀(Chromatin Immunoprecipitation,ChIP)是用来研究蛋白质与DNA是否在体内存在相互作用的一项技术,在表观遗传学中应用十分广泛。该技术主要是利用抗原抗体特异性结合的特性,将与目的蛋白相结合的DNA片段沉淀下来。
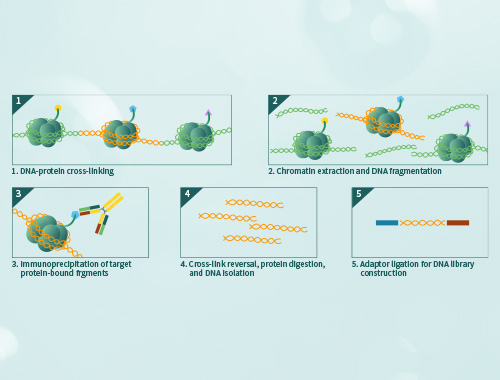
在这个实验中,交联和抗体是影响实验结果的两大因素。前者操作不当会导致超声破碎效果不好从而DNA片段过长,可能会对后续的测序和数据分析产生不利影响,例如测序困难、覆盖度下降从而降低测序的可靠性和分析结果的准确性、数据解读困难等等。而后者,主要影响富集的效率及可用数据的质量。这一步对整个实验来说意义重大,因此抗体的特异性尤为重要。
首先,选择ChIP级别的抗体是做该实验的基础。ChIP相对于IP多出了甲醛交联的过程,同时甲醛会在DNA和蛋白质、蛋白和蛋白的氨基/亚氨基之间形成共价键,ChIP级抗体能识别此状态的目的蛋白。
其次,从特异性角度来说,单克隆抗体识别靶蛋白上的单一表位,相较多克隆抗体通常表现出更高水平的特异性、低非特异性结合和低背景信号。当然,单抗也不是完美的。ChIP实验主要还是体内环境多一些,除了目的蛋白还会存在很多其他染色质相关蛋白,而“交联”这个步骤,可能导致单抗的识别位点被掩蔽,不能有效结合目的蛋白。在这方面,多抗则有更大几率避免这类情况发生。多抗可以识别靶蛋白上的多个表位,即使有些表位被掩蔽,也可以更有效地检测靶标。相对地,多表位也意味着非特异性结合的增多。因此,单抗多抗各有优劣,需要根据实际情况来选择。
那么,从什么渠道找ChIP抗体更迅速、准确、可靠呢?我们给大家总结了三种方式。
01、通过cistrome数据库来查询
cistrome数据库共收录了56464人和小鼠的转录因子、组蛋白修饰和染色质可及性样本,是目前研究ChIP-seq和DNase-seq较为全面的一个数据库。
首先,进入cistrome网站:

以人源转录因子“ARID1A”为例,进行搜索:

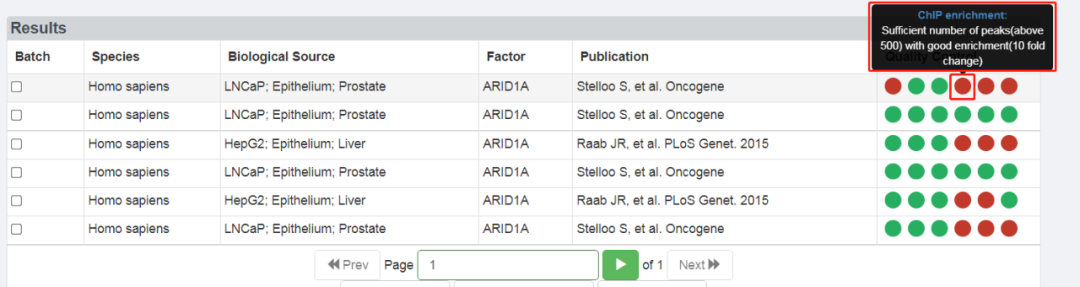
一般来说,绿色越多说明这个实验的数据越可靠,选择全部是绿色的数据进行参考较为有意义。在没有全绿的情况下,可以看代表富集效率的第四项是否为绿色,若是绿色的也可参考。
先选择全绿的一项:
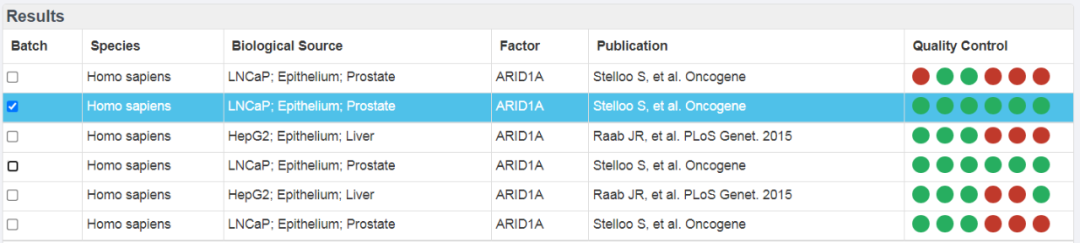
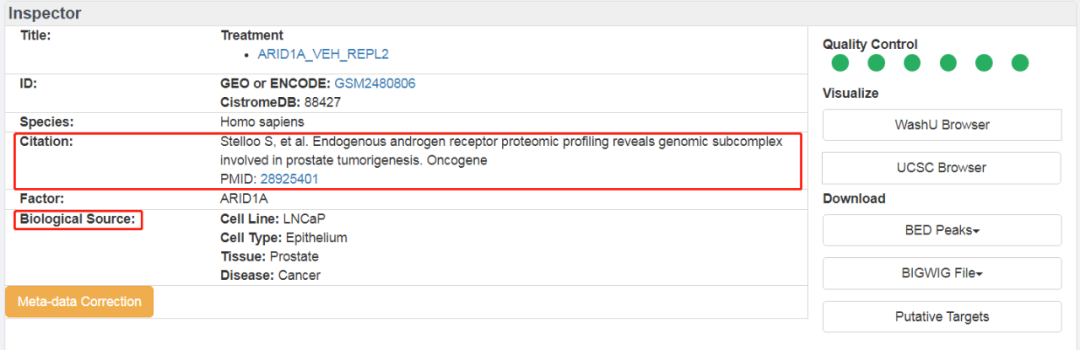
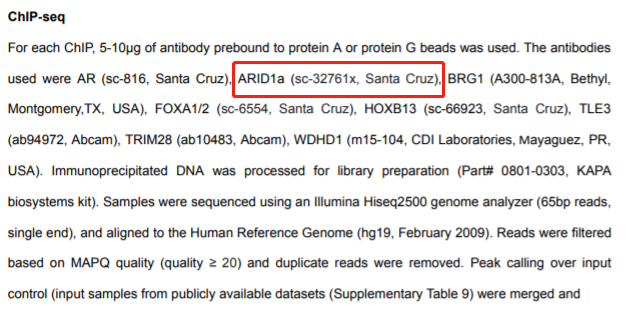
可以看到这个实验数据来源哪一篇文章以及生物样本类型,可跳转到PubMed。材料和方法部分会注明ChIP抗体的货号。
02、通过华美生物、CST等抗体官网搜索
通过华美生物、CST等抗体官网搜索,看产品是否有ChIP应用,及引用的文献数量。在能应用于ChIP实验的基础上,参考的文献越多越有选择的价值。
以人源转录因子“HIST1H1C”为例,进入华美官网。搜索:
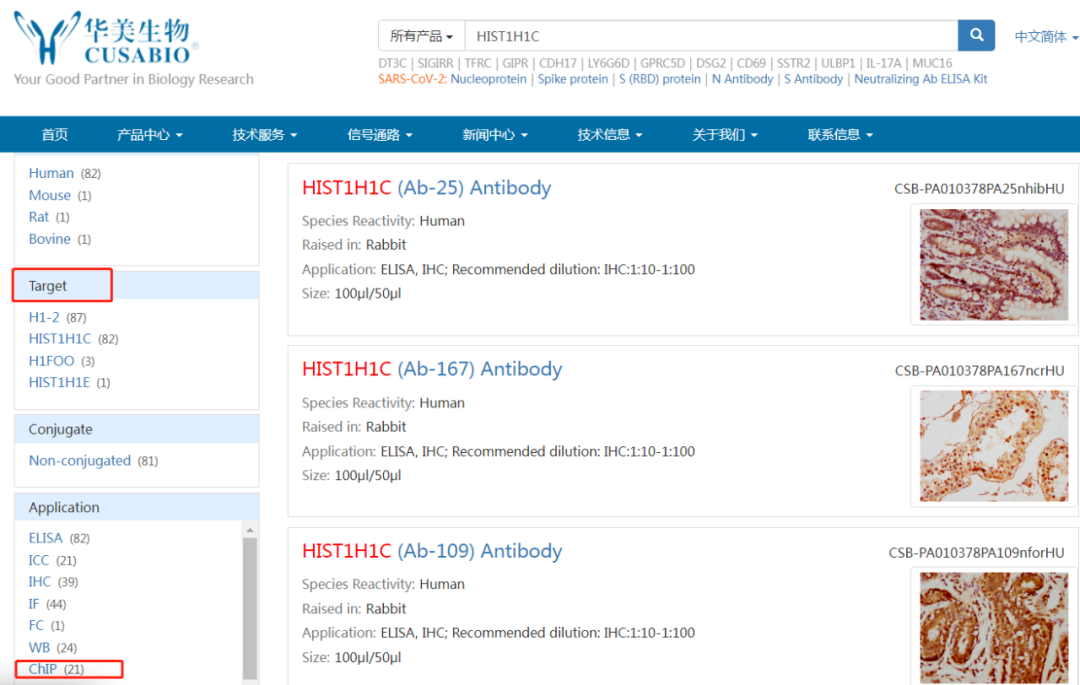

进行筛选后,能看到ChIP抗体数量以及相关文章信息,还可以直接跳转到文章,了解和参考前人的实验思路和结果呈现方式。
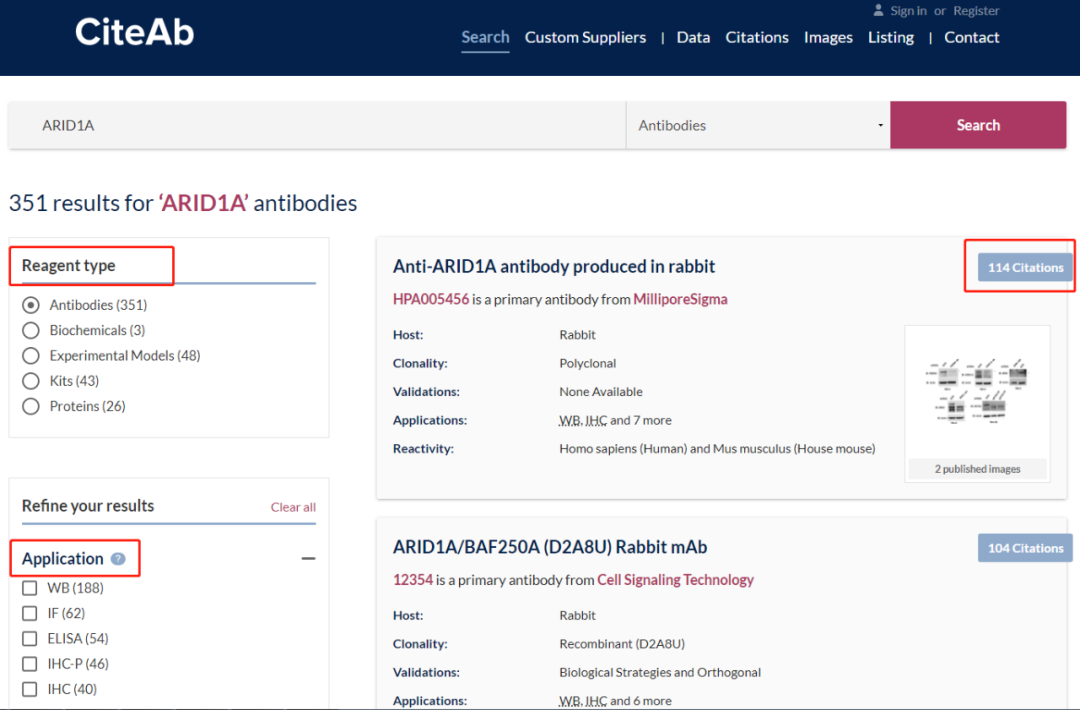
像这样具体到某个抗体官网来进行搜索可能有些麻烦,这里有一个网站CiteAb。其号称是世界最大的抗体搜索引擎,囊括了600+万种抗体、332家试剂及抗体供应商、400+万文献引用;搜索结果可根据抗体被引用的文献次数来排序,对于选择困难症的同学来说可谓是十分方便的一个途径。比起去某个官网查找,这个网站更全面、快捷,十分推荐使用。
03、在文献搜索引擎里,以关键词“目标蛋白+ChIP”进行检索
在文献搜索引擎里,以关键词“目标蛋白+ChIP”进行检索,看文章里的抗体来源。
还是以“ARID1A”为例,进入文献检索平台,输入关键词:

以上,就是找寻ChIP抗体的方法。简而言之,尽量选择有文献支撑的抗体来使用。
当然了,也不是所有的蛋白都有ChIP抗体。实在是没有针对目标蛋白的ChIP抗体时,也可以考虑从标签抗体入手。不过标签抗体的特异性不是很高,富集效果可能没有特异性抗体好,这一点需要提前考虑到。若不想选择标签抗体,也可以退而求其次,选择针对目标蛋白的IP、IF级别的抗体。
参考文献:
[1]Immunoprecipitation.Cold Spring Harb Protoc; 2020; doi:10.1101/pdb.top098509
[2]Zheng R, Wan C, Mei S, Qin Q, Wu Q, Sun H, Chen CH, Brown M, Zhang X, Meyer CA, Liu XS. Cistrome Data Browser: expanded datasets and new tools for gene regulatory analysis. Nucleic Acids Res; 2018 ; doi: 10.1093/nar/gky1094
[3]Mei S, Qin Q, Wu Q, Sun H, Zheng R, Zang C, Zhu M, Wu J, Shi X, Taing L, Liu T, Brown M, Meyer CA, Liu XS. Cistrome data browser: a data portal for ChIP-Seq and chromatin accessibility data in human and mouse. Nucleic Acids Res; 2017; doi: 10.1093/nar/gkw983
[4]Stelloo S, Nevedomskaya E, Kim Y, Hoekman L, Bleijerveld OB, Mirza T, Wessels LFA, van Weerden WM, Altelaar AFM, Bergman AM, Zwart W. Endogenous androgen receptor proteomic profiling reveals genomic subcomplex involved in prostate tumorigenesis. Oncogene. 2018; doi: 10.1038/onc.2017.330
[5]Qu, L, Deng, C, Luo Q, Shang X, Wu Jiao, Shi Y, Wang L, Han Z.regulates insulin sensitivity and lipid metabolism.eBioMedicine.2019; doi:10.1016/j.ebiom.2019.03.021

最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










