史上最全的ATAC-seq实验技术详解
ATAC-seq(Assay for Transposase-Accessible Chromatin using sequencing)是一种通过高通量测序技术检测染色质开放区域的方法。使用改造的 Tn5转座酶(携带测序接头),靶向插入基因组中开放的染色质区域(即未被核小体紧密包裹的DNA),同时完成DNA的片段化和测序接头的标记,将经转座酶处理的DNA片段通过PCR扩增,构建测序文库并进行高通量测序。
ATAC-seq通过绘制全基因组染色质可及性图谱,揭示细胞类型特异的表观遗传调控机制,目前已广泛应用于发育生物学、疾病研究(如癌症异质性)和药物靶点筛选等领域。
一、技术应用
1.初步判断所研究的课题是否涉及表观遗传调控机制
2.结合基序分析,识别参与基因调控的转录因子
3.识别转录因子调控的靶基因和功能元件
4.与超级增强子鉴定联合分析,明确活性超级增强子的范围
5.更好地了解药物或疾病的基因调控和细胞应答机制
6.识别驱动细胞命运、疾病或应答相关的转录因子
二、技术优势
1、高灵敏度: ATAC-seq可以检测到基因组中极少量的可及染色质区域,使其在样本资源有限的情况下也能获得有意义的结果。
2、低样本需求: 由于ATAC-seq需要的细胞数较少,特别是与其他染色质可及性测序技术相比,可在稀有细胞样本中应用。
3、高通量测序: 使用高通量测序技术,ATAC-seq能够同时分析数百万个片段,提供全面的基因组范围数据。
4、直接检测开放染色质: 通过标记开放染色质区域,ATAC-seq直接反映了基因组的可及性,而不需要附加步骤。
5、快速实验流程: 相比传统的染色质可及性测序方法,ATAC-seq的实验流程简化,减少了样本处理时间。
6、较少的偏差: 由于ATAC-seq使用转座酶进行标记,相对于其他方法,它具有较少的偏差,更好地捕获染色质区域的可及性。
7、分辨率高: ATAC-seq可以提供较高的基因组分辨率,更精确地标记和定位开放染色质区域。
8、可提供与其他组学联合分析思路;提供个性化分析方案。
三、ATAC-seq实验流程

01.细胞核提取
收集目标细胞(新鲜或快速冷冻,确保细胞活性不低于90%,细胞活性至关重要!死细胞会导致背景噪音)。
细胞核提取:将细胞或组织样本加入裂解液,冰上孵育使细胞膜通透,然后离心去除上清,加入PBS重悬细胞核。
细胞核计数:取适量细胞核悬液,加入台盼蓝溶液染色,用血球计数板在显微镜下计数,计算细胞核浓度。
02.转座酶片段化
配置反应体系,将透化后的细胞核与预加载了接头的Tn5转座酶混合。在适宜温度(通常37°C)下孵育(30-60分钟)。Tn5转座酶优先结合并插入到染色质结构开放、无核小体占据或核小体疏松的区域。在插入位点两侧切割DNA双链。同时将预加载的测序接头连接到被切割的DNA片段两端。
03.DNA提取与PCR扩增
DNA提取:使用DNA富集beads进行全回收,80%乙醇清洗两遍,晾干2min左右,避免磁珠过于干燥导致回收率偏低,然后溶水回收
PCR扩增:使用包含测序平台(如Illumina)所需完整接头的引物进行PCR扩增。
04.文库构建
冰上配制扩增mix,主要为测序引物Index和扩增酶, PCR引物上带有独特的样本索引序列,使得多个样本的文库可以在同一个测序通道中进行混合测序,之后通过索引区分样本。
再次纯化:PCR后使用磁珠纯化扩增产物,去除引物二聚体等杂质。
使用高灵敏度仪器定量和质控文库,检测文库浓度和片段大小分布是否符合预期。
05.高通量测序
目的:对文库中的DNA片段进行大规模并行测序,获得片段两端的序列信息。
建议使用配对末端测序,可以获得更多的序列数据带来更好的比对结果,PCR重复的识别更加准确。
测序深度:取决于参考基因组的大小和预期的开放染色质程度
一般推荐:常规细胞系/组织样本:30-50 Million (M) 有效比对 reads
稀有细胞类型/单细胞ATAC数据整合:可能需要更深(>50M)
单细胞ATAC-seq:每个细胞通常几千到几万条reads,但总reads量巨大
四、金开瑞ATAC-seq实验数据分析
◆数据基本处理与质控:对原始数据进行过滤,去除接头信息、低质量碱基和未测出的碱基,获取有效数据(Clean Data)
◆文库插入片段长度分布
◆基因组测序深度累积分布
◆各样品基因promoter(up2K)区测序深度分布
◆Peak检测
◆Peak长度分布
◆Peak深度分布
◆样本生物学重复 IDR 分析
◆Peak在基因功能元件上的分布
◆Peak相关基因
◆Peak相关基因的GO功能显著性富集分析
◆Peak相关基因的pathway富集分析
◆样本间差异peak检测
◆样品间差异peak在基因功能元件的分布
◆样品间差异peak相关基因
◆样品间差异peak相关基因的GO和KEGG富集分析
五、案例分析
这篇文章发表在Nature Plants(IF:15.8),通过多组学策略(ATAC-seq、RNA-seq、CUT&Tag)揭示染色质动态与基因表达的协同作用。并确定了关键转录因子和潜在的转化效率增强子。
为了连接转录因子和靶基因,研究者使用 ATAC-seq 数据确定了足迹。大多数足迹分布在 ATAC-seq 峰的中间,宽度为 10 - 50 个碱基对,其中一半可以与拟南芥的转录因子结合基序相匹配。正如预期的那样,足迹的序列比基因间区域的序列更保守。对差异足迹的分析显示,参与植物再生的已知调节因子(如 LEC2、DOF5.6、WOX11 和 BBM)的转录因子活性发生了显著变化,表明它们潜在的功能重要性。
为了确定基因表达的时间顺序,研究者进行了拟时间分析,发现转录因子和靶基因在再生过程中表现出相似的转录谱。每个聚类中的转录因子调节同一聚类中的靶基因。在 C1(在第 0 天特别高表达)与其他聚类之间没有观察到调控关系,这进一步强调了生长素处理的影响。有趣的是,发现 C5 - C6 - C2 - C3 - C4 聚类之间的调控关系遵循基因表达的时间线。
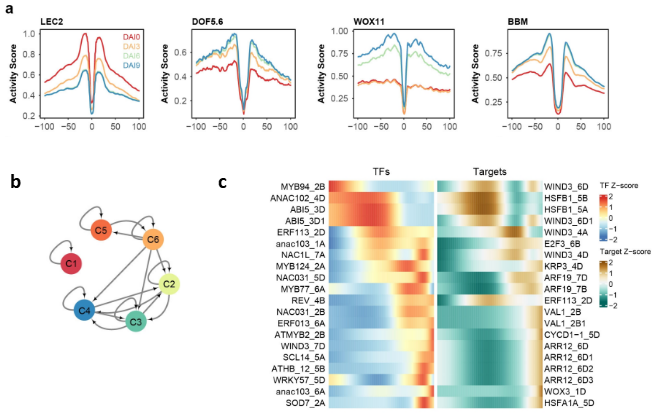
Figure a: 在不同感应阶段的LEC2,DOF5.6,WOX11和BBM的ATAC-SEQ足迹;活动评分反映了染色质的可及性。
Figure 4 b:不同的RNA簇之间的调节关系(Fisher的精确测试,p.adj < 1e-6))。
Figure 4c:TRN中TF和目标的表达模式, TFS与目标之间的调节关系已在拟南芥中证明。(来源于文献:Uncovering the transcriptional regulatory network involved in boosting wheat regeneration and transformation. Nat Plants)
ATAC-seq是一项革命性的技术,以其简便、灵敏、高分辨的特点,成为绘制染色质可及性图谱、解析基因调控网络的强大工具它在基础生物学、发育、疾病机制和精准医疗等领域发挥着越来越重要的作用。该技术无需抗体或复杂交联步骤,显著节省时间成本,而且可精准定位开放染色质区域(分辨率达单碱基水平),识别转录因子结合位点及核小体排布,适用于微量样本(低至500~50,000个细胞),尤其适合珍贵临床样本或单细胞分析。金开瑞可以提供ATAC-seq、ChIP-seq和CUT&Tag技术服务,期待与您携手合作,共同推动生命科学的发展。
最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










