DLO-HiC三维基因组
每个人体内都有着两米长的DNA,它紧密折叠在直径10微米小的细胞核内。基因组的三维空间结构对基因组的表达、调控等功能有重要的影响。
基因组三维空间结构与功能的研究简称三维基因组学(Three-Dimensional Genomics, 3D Genomics),指在考虑基因组序列、基因结构及其调控元件的同时,对基因组序列在细胞核内的三维空间结构,及其对基因转录、复制、修复和调控等生物过程中功能的研究。
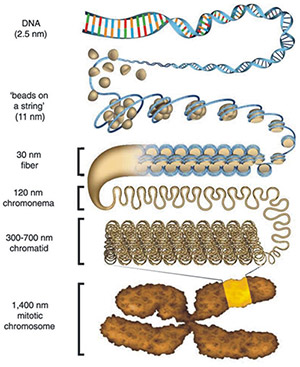
细胞核中染色质折叠模型示意图
(引自 Ou HD, Phan S, Deerinck TJ, Thor A, Ellisman MH, O'Shea CC. ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 2017;357(6349):eaag0025.)
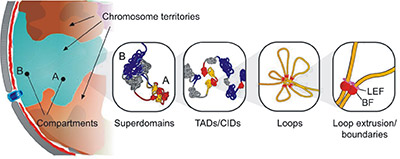
细胞核中染色质三维空间结构分解
(引自 Pueschel R, Coraggio F, Meister P. From single genes to entire genomes: the search for a function of nuclear organization[J]. Development, 2016 , 143 (6) :910.)
2003 年,Job Dekker提出了染色质构象捕获技术(Chromatin Conformation Capture, 3C),用于测定特定的点到点之间的染色质交互作用。2006年,在3C技术基础上,Simonis M开发了4C技术(Circularized Chromatin Conformation Capture),用于测定一点到多点之间的染色质交互作用。Dostie等人研发了5C技术(Carbon-Copy Chromatin Conformation Capture),用于测定多点到多点之间的染色质交互作用。
为了能捕获全基因组范围的染色质相互作用,Job Dekker 开发出 Hi-C 技术,并将其应用于揭示染色质的三维结构中。Hi-C将线性距离远、空间结构近的DNA片段进行交联, 并富集交联的DNA片段进行高通量测序,通过测序数据分析揭示染色质的远程相互作用,推导基因组的三维空间结构和可能的基因间的调控关系。
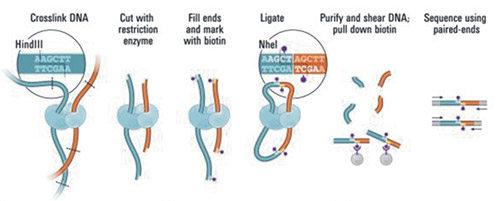
Hi-C 实验流程示意图
(引自 Lieberman-Aiden E, van Berkum NL, et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome[J]. Science. 2009, 326(5950):289-93.)
Hi-C 技术可用于研究:1.染色质的层级结构(A/B compartments、TADs、Loops);2. 关联基因组上原本分散的远距离调控元件与其具体调控区域;3. 解析基因的转录调控、增强子与启动子的作用机制;4. 协助发现疾病易感位点、DNA 损伤修复、基因组结构变异和表观遗传。
武汉金开瑞生物工程有限公司技术顾问、华中农业大学教授曹罡课题组与华中农业大学李国亮教授课题组在国际著名期刊Nature Genetic( IF=38.33 )上联合发表题为“Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture”的研究论文。该论文主要介绍了一种新的染色质构象捕获技术(Digestion-ligation-only Hi-C, DLO Hi-C),此技术信噪比高,能在早期进行质量控制,为解析基因组三维结构提供了一种新型、高效、经济的研究方法。
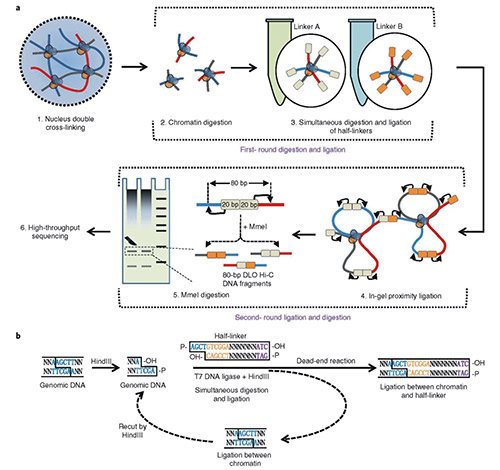
DLO Hi-C 染色质构象捕获技术设计了巧妙的酶切位点,采用同时酶切酶连的方式,将 DNA 接头连接在染色质内切酶切口末端上,然后进行邻近酶连,最后再用 MmeI 内切酶酶切消化,回收固定大小互作 DNA 片段。
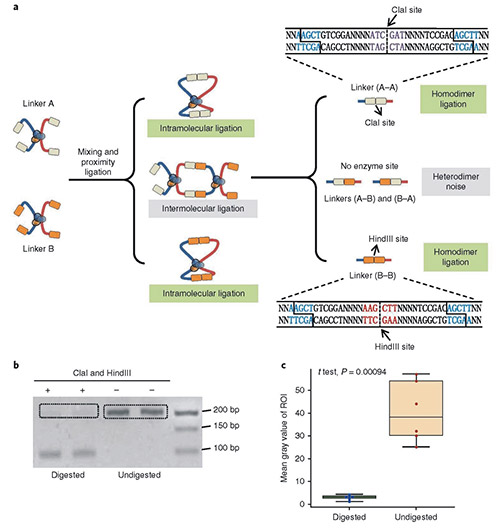
消化后的染色质与两种半接头(接头 A 或接头 B)连接,如果发生分子内连接,那么接头连接后(A-A/B-B)就会形成HindⅢ或ClaI酶切位点,而随机的分子间连接形成的噪音异二聚体(A-B/B-A)上没有限制酶切割位点。
DLO Hi-C 染色质构象捕获技术,具有以下优点:
1. 图谱分辨率高于传统 Hi-C 技术、DNase Hi-C 技术;
2. DLO Hi-C 简化 Hi-C 复杂的文库构建过程;
3. A/B compartments 数据、TADs 数据和 Loops 数据较优于其他 Hi-C 的衍生技术。
RUNX1-mediated alphaherpesvirus-host trans-species chromatin interaction promotes viral transcription. 期刊:Sci Adv. IF:14.14 发表时间:2021
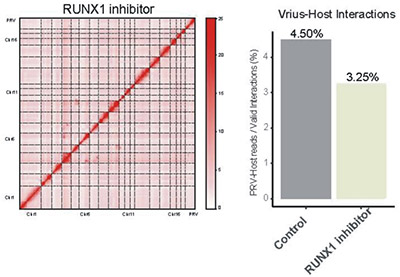
使用伪狂犬病病毒 (PRV) 作为模型病毒,对感染后猪肾PK15细胞系进行了染色体构象捕获测定,以证明病毒和宿主之间的全基因组特异性跨物种染色质相互作用。发现了PRV 基因组由宿主 DNA 结合蛋白 RUNX1 传递到开放染色质和活跃转录区。本研究使用了4C、DLO Hi-C、ChIP-seq、ATAC-seq、RNA-seq、DNA FISH等多种技术。
3D genome architecture coordinates trans and cis regulation of differentially expressed ear and tassel genes in maize. 期刊:Genome Biol. IF:10.81 发表时间:2020
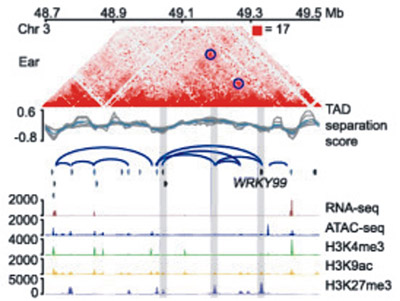
使用 ATAC-seq 和ChIP-seq在玉米穗和流苏发育过程中识别出56,055和52,633个开放染色质区域(OCR),进一步使用DLO Hi-C在相应组织中以高分辨率系统地绘制染色质相互作用。连接OCR和基因的广泛染色质环为负责穗和流苏特异性基因表达的顺式和反式调节元件提供了3D视图。
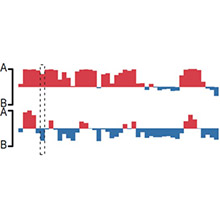

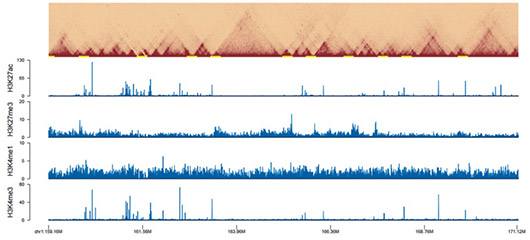
Hi-C与组蛋白修饰ChIP-seq联合分析
| 项目 | 内容 |
| 建库、测序 | 每个样本生成2个文库,每个文库120GB测序量,每个样品产生不少于200GB raw data |
| 标准分析 | 质量控制、染色体顺式、反式互作,全基因组交互、A/B compartment分析、TAD分析 |
| 样本组间差异A/B、TADs、Loops比对 | 商议 |
| Hi-C与RNA联合分析 | |
| 与其他组学联合分析 |
最新动态
-
01.29
技术跨界联用:表观遗传学进入“多维验证”时代,这些疾病机制再也藏不住了!文末有惊喜活动!
-
01.29
机制研究的金标准:金开瑞RIP/RNA pull-down试剂盒获多篇顶刊研究认可!
-
01.29
想测结合常数,但样本太珍贵?来试试MST技术
-
01.29
双荧光素酶实验,如何助力客户连发IF>10高分文章?揭秘我们的核心技术与成功案例!
-
01.29
传统草药新生命:鱼腥草来源外囊泡或成脑卒中治疗“绿色疗法”
-
01.29
FRONT PHARMACOL | 三七外泌体通过调控PI3K/AKT信号通路促进大鼠骨髓间充质干细胞的成骨分化
-
12.24
导师压箱底的DNA pull down实验全流程拆解!
-
12.24
从靶点发现到药物验证:SPR +CoIP +ChIP+双荧光+pull down共同验证雷公藤红素如何为胰腺癌提供全新治疗线索?
-
12.24
REGEN BIOMATER| 首次揭示-红景天来源纳米囊泡在血管再生治疗中的突破性进展!
-
12.10
文献阅读:一分钟教你看懂火山图










